













| General Information: |
|
| Name(s) found: |
HSP104 /
YLL026W
[SGD]
|
| Description(s) found:
Found 26 descriptions. SHOW ALL |
|
| Organism: | Saccharomyces cerevisiae |
| Length: | 908 amino acids |
Gene Ontology: |
|
| Cellular Component: |
nucleus
[IDA cytoplasm [IDA |
| Biological Process: |
telomere maintenance
[IMP chaperone mediated protein folding requiring cofactor [IDA protein refolding [IDA response to stress [IDA |
| Molecular Function: |
chaperone binding
[IDA ATPase activity, coupled [IDA |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MNDQTQFTER ALTILTLAQK LASDHQHPQL QPIHILAAFI ETPEDGSVPY LQNLIEKGRY 60
61 DYDLFKKVVN RNLVRIPQQQ PAPAEITPSY ALGKVLQDAA KIQKQQKDSF IAQDHILFAL 120
121 FNDSSIQQIF KEAQVDIEAI KQQALELRGN TRIDSRGADT NTPLEYLSKY AIDMTEQARQ 180
181 GKLDPVIGRE EEIRSTIRVL ARRIKSNPCL IGEPGIGKTA IIEGVAQRII DDDVPTILQG 240
241 AKLFSLDLAA LTAGAKYKGD FEERFKGVLK EIEESKTLIV LFIDEIHMLM GNGKDDAANI 300
301 LKPALSRGQL KVIGATTNNE YRSIVEKDGA FERRFQKIEV AEPSVRQTVA ILRGLQPKYE 360
361 IHHGVRILDS ALVTAAQLAK RYLPYRRLPD SALDLVDISC AGVAVARDSK PEELDSKERQ 420
421 LQLIQVEIKA LERDEDADST TKDRLKLARQ KEASLQEELE PLRQRYNEEK HGHEELTQAK 480
481 KKLDELENKA LDAERRYDTA TAADLRYFAI PDIKKQIEKL EDQVAEEERR AGANSMIQNV 540
541 VDSDTISETA ARLTGIPVKK LSESENEKLI HMERDLSSEV VGQMDAIKAV SNAVRLSRSG 600
601 LANPRQPASF LFLGLSGSGK TELAKKVAGF LFNDEDMMIR VDCSELSEKY AVSKLLGTTA 660
661 GYVGYDEGGF LTNQLQYKPY SVLLFDEVEK AHPDVLTVML QMLDDGRITS GQGKTIDCSN 720
721 CIVIMTSNLG AEFINSQQGS KIQESTKNLV MGAVRQHFRP EFLNRISSIV IFNKLSRKAI 780
781 HKIVDIRLKE IEERFEQNDK HYKLNLTQEA KDFLAKYGYS DDMGARPLNR LIQNEILNKL 840
841 ALRILKNEIK DKETVNVVLK KGKSRDENVP EEAEECLEVL PNHEATIGAD TLGDDDNEDS 900
901 MEIDDDLD |
| PUBLICATION | TOPOLOGY | COCOMPLEXED PROTEINS | |
| View Details | Riffle et al. (2010) (Unpublished Data) |
|
ABP1 CAM1 EFB1 FBA1 HSC82 HSP104 HSP82 HYP2 SSZ1 TEF4 TIF1, TIF2 YEF3 ZUO1 |
| View Details | Krogan NJ, et al. (2006) |

|
AHA1 BUD3 CNS1 CPR6 DIG2 DNM1 DSF1, YNR073C FRT2 GPA2 HOM6 HSC82 HSP104 HSP82 MBB1 MRPL50 PAM1 PPT1 QNS1 RMD8 RTS1 SMT3 STI1 STP4 TDH1 UTP20 YBL104C |
| View Details | Ho Y, et al. (2002) |

|
ABP1 ACO1 ADE5,7 ADH2 AHA1 AIM41 ATP4 BCY1 BUD14 CAR2 CMP2 COR1 CPR6 CRZ1 CYS4 DCP1 DCP2 DNM1 DOG1 ECM10 EDC3 EDE1 EGD2 ENP1 ESS1 GAS1 GCN3 GIS4 GND1 GPH1 HEF3 HHF1, HHF2 HRR25 HSP104 HXT6 HXT7 HYP2 IPP1 KEL1 KEL2 KGD1 KIN2 KRE6 LOC1 LSP1 LTV1 MDH1 MDN1 MDS3 MGE1 NAN1 NSA1 NTA1 NUG1 OXP1 OYE2 PEX19 PFK1 PIL1 PIN4 POP2 PTC4 PUF3 PUP2 RFC1 RFC2 RFC4 RIO2 RPB3 RPC19 RPM2 RPN1 RPO21 RPT3 SAP185 SAP190 SAS10 SCS2 SEC2 SEC23 SES1 SFB3 SGM1 SIT4 SPT5 TEF4 TFC4 TFG1 TFP1 TMA19 TOM1 TPS2 TSR1 UBA1 UTP10 UTP22 UTP9 VMA4 YBR225W YER138C YHR020W YHR033W |
The following runs contain data for this protein:
| BAIT | COMMENTS | PUBLICATION | |
| View Run | #12 Mitotic Prep1-TiO2 Flowthrough | Keck JM, et al. (2011) |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
| PROTEIN(S) | PUBLICATION | |
| View Data |
|
Huh WK, et al. (2003) |
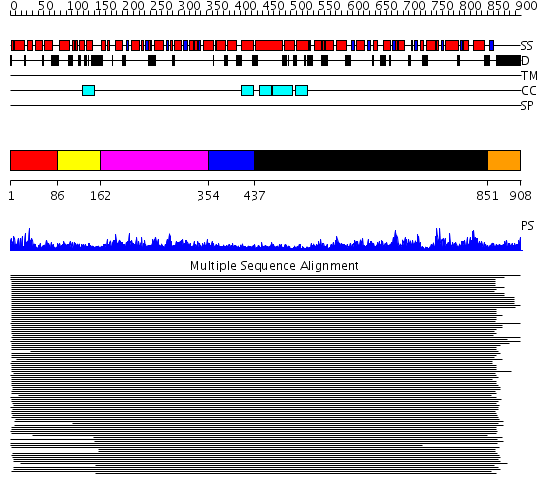
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..85] | 3.167994 | View MSA. Confident ab initio structure predictions are available. | |
| 2 | View Details | [86..161] | 9.366532 | Clp amino terminal domain No confident structure predictions are available. | |
| 3 | View Details | [162..353] | 679.69897 | ClpB | |
| 4 | View Details | [354..436] | 9.02 | N-terminal, ClpS-binding domain of ClpA, an Hsp100 chaperone; ClpA, an Hsp100 chaperone, AAA+ modules | |
| 5 | View Details | [437..850] | 6.0 | HslU | |
| 6 | View Details | [851..908] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.94 |
Source: Reynolds et al. (2008)