













| General Information: |
|
| Name(s) found: |
PHO85 /
YPL031C
[SGD]
|
| Description(s) found:
Found 31 descriptions. SHOW ALL |
|
| Organism: | Saccharomyces cerevisiae |
| Length: | 305 amino acids |
Gene Ontology: |
|
| Cellular Component: |
nucleus
[IDA |
| Biological Process: |
glycogen metabolic process
[IDA response to drug [IMP telomere maintenance [IMP protein amino acid phosphorylation [IDA phosphate metabolic process [TAS cell cycle [IGI |
| Molecular Function: |
cyclin-dependent protein kinase activity
[IDA |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSSSSQFKQL EKLGNGTYAT VYKGLNKTTG VYVALKEVKL DSEEGTPSTA IREISLMKEL 60
61 KHENIVRLYD VIHTENKLTL VFEFMDNDLK KYMDSRTVGN TPRGLELNLV KYFQWQLLQG 120
121 LAFCHENKIL HRDLKPQNLL INKRGQLKLG DFGLARAFGI PVNTFSSEVV TLWYRAPDVL 180
181 MGSRTYSTSI DIWSCGCILA EMITGKPLFP GTNDEEQLKL IFDIMGTPNE SLWPSVTKLP 240
241 KYNPNIQQRP PRDLRQVLQP HTKEPLDGNL MDFLHGLLQL NPDMRLSAKQ ALHHPWFAEY 300
301 YHHAS |
| PUBLICATION | TOPOLOGY | COCOMPLEXED PROTEINS | |
| View Details | Riffle et al. (2010) (Unpublished Data) |
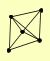
|
PCL7 PCL8 PHO80 PHO81 PHO85 |
| View Details | (MIPS) Mewes HW, et al. (2004) |
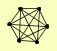
|
PCL1 PCL10 PCL2 PCL8 PHO80 PHO85 |
| View Details | Krogan NJ, et al. (2006) |
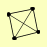
|
MMR1 PCL6 PCL8 PHO85 |
| View Details | Ho Y, et al. (2002) |
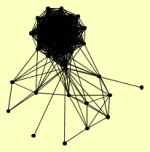
|
ADK1 AGX1 ATP3 AVO1 CCT5 CLU1 DHH1 DPB2 FUN12 GAL7 GLC7 GLC8 GPH1 IDH1 ILV2 MET16 MKK2 NOG2 NPA3 PCL6 PCL7 PDX1 PHO81 PHO85 PPZ2 PUF3 RIM15 RPN12 SAT4 SIS1 SLX1 SOD1 SRP54 STI1 TEM1 TFC7 VMA8 YER077C YGR266W YHB1 YKR051W YKU80 YLR271W YPR003C |
The following runs contain data for this protein:
| BAIT | COMMENTS | PUBLICATION | |
| View Run | No Comments | McCusker D, et al (2007) | |
| View Run | sample cbf3 from feb 2005 | Sandall S, et all (2006) | |
| View Run | Sample 2- cpn2 from june 2005 | McCusker D, et al (2007) | |
| View Run | Sample 3 - cpn + atp, from june 2005 | McCusker D, et al (2007) | |
| View Run | #03 Alpha Factor Prep1-TiO2 Flowthrough | Keck JM, et al. (2011) |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
| PROTEIN(S) | PUBLICATION | |
| View Data |
|
Huh WK, et al. (2003) |
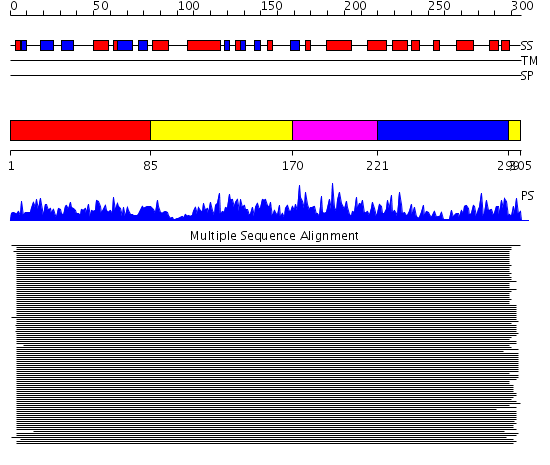
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..84] | 1080.218487 | Cyclin-dependent PK, CDK2 | |
| 2 | View Details | [85..169] [299..305] |
1080.218487 | Cyclin-dependent PK, CDK2 | |
| 3 | View Details | [170..220] | 1080.218487 | Cyclin-dependent PK, CDK2 | |
| 4 | View Details | [221..298] | 1080.218487 | Cyclin-dependent PK, CDK2 |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.97 |
Source: Reynolds et al. (2008)