













| General Information: |
|
| Name(s) found: |
CBK1 /
YNL161W
[SGD]
|
| Description(s) found:
Found 25 descriptions. SHOW ALL |
|
| Organism: | Saccharomyces cerevisiae |
| Length: | 756 amino acids |
Gene Ontology: |
|
| Cellular Component: |
nucleus
[IDA cellular bud [IDA cytoplasm [IDA cellular bud neck [IDA mating projection tip [TAS |
| Biological Process: |
response to pheromone involved in conjugation with cellular fusion
[IDA cell morphogenesis during vegetative growth [IGI regulation of exit from mitosis [IMP cell morphogenesis involved in conjugation with cellular fusion [IDA |
| Molecular Function: |
protein kinase activity
[ISS |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MYNSSTNHHE GAPTSGHGYY MSQQQDQQHQ QQQQYANEMN PYQQIPRPPA AGFSSNYMKE 60
61 QGSHQSLQEH LQRETGNLGS GFTDVPALNY PATPPPHNNY AASNQMINTP PPSMGGLYRH 120
121 NNNSQSMVQN GNGSGNAQLP QLSPGQYSIE SEYNQNLNGS SSSSPFHQPQ TLRSNGSYSS 180
181 GLRSVKSFQR LQQEQENVQV QQQLSQAQQQ NSRQQQQQLQ YQQQQQQQQQ QQHMQIQQQQ 240
241 QQQQQQQQSQ SPVQSGFNNG TISNYMYFER RPDLLTKGTQ DKAAAVKLKI ENFYQSSVKY 300
301 AIERNERRVE LETELTSHNW SEERKSRQLS SLGKKESQFL RLRRTRLSLE DFHTVKVIGK 360
361 GAFGEVRLVQ KKDTGKIYAM KTLLKSEMYK KDQLAHVKAE RDVLAGSDSP WVVSLYYSFQ 420
421 DAQYLYLIME FLPGGDLMTM LIRWQLFTED VTRFYMAECI LAIETIHKLG FIHRDIKPDN 480
481 ILIDIRGHIK LSDFGLSTGF HKTHDSNYYK KLLQQDEATN GISKPGTYNA NTTDTANKRQ 540
541 TMVVDSISLT MSNRQQIQTW RKSRRLMAYS TVGTPDYIAP EIFLYQGYGQ ECDWWSLGAI 600
601 MYECLIGWPP FCSETPQETY RKIMNFEQTL QFPDDIHISY EAEDLIRRLL THADQRLGRH 660
661 GGADEIKSHP FFRGVDWNTI RQVEAPYIPK LSSITDTRFF PTDELENVPD SPAMAQAAKQ 720
721 REQMTKQGGS APVKEDLPFI GYTYSRFDYL TRKNAL |
The following runs contain data for this protein:
| BAIT | COMMENTS | PUBLICATION | |
| View Run | No Comments | Hazbun TR, et al. (2003) | |
| View Run | his-HA tag on RPA135 | Schneider, DA, et al. (2006) | |
| View Run | his-HA tag on RPA135 | Schneider, DA, et al. (2006) | |
| View Run | #12 Mitotic Prep1-TiO2 Flowthrough | Keck JM, et al. (2011) | |
| View Run | #30 Asynchronous Prep6-TiO2 Phosphopeptide enrichment | Keck JM, et al. (2011) | |
| View Run | #11 Mitotic Prep1-TiO2 enriched, new search criteria | Keck JM, et al. (2011) | |
| View Run | #17 Mitotic Prep3-TiO2 Phosphopeptide enrichment | Keck JM, et al. (2011) |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
| PROTEIN(S) | PUBLICATION | |
| View Data |
|
Huh WK, et al. (2003) |
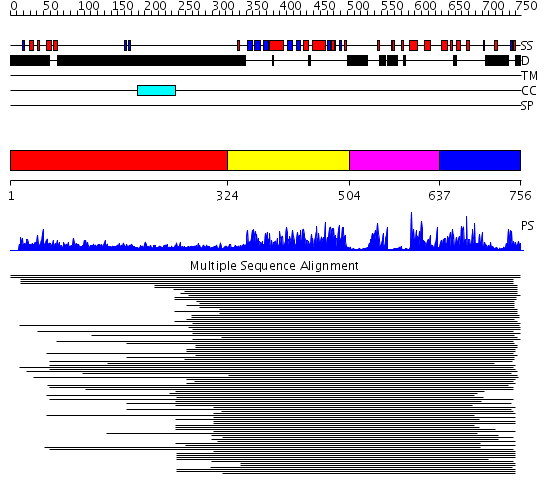
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..323] | N/A | No confident structure predictions are available. | |
| 2 | View Details | [324..503] | 94.0 | No description for 1lhxA_ was found. | |
| 3 | View Details | [504..636] | N/A | No confident structure predictions are available. | |
| 4 | View Details | [637..756] | 440.228787 | cAMP-dependent PK, catalytic subunit |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.98 |
Source: Reynolds et al. (2008)