













| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MVSWGRGKDA YYLYISREQE EDDDDSLSFY SSQKDTEDEF CGCLFPDPEI PGSSSSSGCS 60
61 SSSTELYDLA AAHAALISRQ QQILSQAIPI IPEHQLAAVA AHHQHHQQLH PSVQYQLVAA 120
121 ATHHNHHQPQ AAQPHYSAVV PRSDVIQQPP HFALHHHLQN LVQQQQQQQA HHHHQQLVGE 180
181 MALVSHTHPA AVGSTTCYEK NQQKQQQVQQ IPTQPQVAHV SSNAILAAAQ PFYPPPVQDS 240
241 QPDRPIGYGA FGVVWSVTDP RSGKRVALKK MPNVFQNLAS CKRVFREIKM LSSFRHDNVL 300
301 SLLDILQPAN PSFFQELYVL TELMQSDLHK IIVSPQALTP DHVKVFVYQI LRGLKYLHTA 360
361 NILHRDIKPG NLLVNSNCIL KICDFGLART WDQRDRLNMT HEVVTQYYRA PELLMGARRY 420
421 TGAVDIWSVG CIFAELLQRK ILFQAAGPIE QLQMIIDLLG TPSQEAMKYA CEGAKNHVLR 480
481 AGLRAPDTQR LYKIASPDDK NHEAVDLLQK LLHFDPDKRI SVEEALQHRY LEEGRLRFHS 540
541 CMCSCCYTKP NMPSRLFAQD LDPRHESPFD PKWEKDMSRL SMFELREKMY QFVMDRPALY 600
601 GVALCINPQS AAYKNFASSS VAQASELPPS PQAW |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
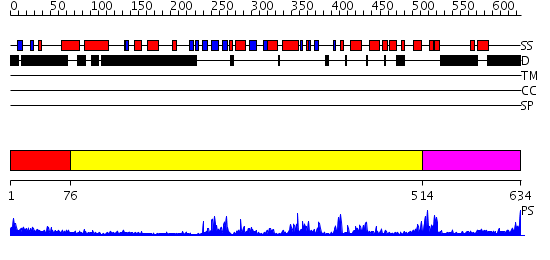
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..75] | N/A | No confident structure predictions are available. | |
| 2 | View Details | [76..513] | 47.30103 | No description for 2fo0A was found. | |
| 3 | View Details | [514..634] | 85.69897 | No description for 2gphA was found. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.98 |
Source: Reynolds et al. (2008)