













| General Information: |
|
| Name(s) found: |
sid2 /
SPAC24B11.11c
[Sanger Pombe]
|
| Description(s) found:
Found 21 descriptions. SHOW ALL |
|
| Organism: | Schizosaccharomyces pombe |
| Length: | 607 amino acids |
Gene Ontology: |
|
| Cellular Component: |
medial cortex
[IDA spindle pole body [IDA Sid2-Mob1 complex [IPI cytosol [IDA |
| Biological Process: |
regulation of cytokinesis
[IMP protein amino acid phosphorylation [ISS septation initiation signaling [IMP |
| Molecular Function: |
ATP binding
[ISS protein binding [IPI protein serine/threonine kinase activity [TAS |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MNRVNDMSPV EGDLGLQLSS EADKKFDAYM KRHGLFEPGN LSNNDKERNL EDQFNSMKLS 60
61 PVASSKENYP DNHMHSKHIS KLPIASPIPR GLDRSGELSY KDNNHWSDRS STGSPRWENG 120
121 SMNLSVEEME KVVQPKVKRM ATICQMFFLD HYFEQLHYLY TRKQRARLFE EQLLKEPDSR 180
181 RDELVKRYNG RERVYLRKRR TRISHGDFQT ITQVGQGGYG SVWLARKRDT KEIVALKIMN 240
241 KSVLHKMDEI RHVLTERDIL TTANSEWLVR LLYAFQDTSN IYLAMEFVPG GDFRTLLSNS 300
301 GVLRDHHAKF YATEMFLAID ALHQLGYIHR DLKPENFLVG ASGHIKLTDF GLSSGIISKK 360
361 KIESMKIRLQ EVNNVVVPER SMRERRQVFR TLLSQDPVYA HSVVGSPDYM APEVLRGENY 420
421 NHSVDYWSLG CIMYECLSGF PPFSGSNVNE TWSNLKNWRK CFQRPHYDDP RDLEFNWRDD 480
481 AWDFVCHCIT DPKDRFCSLK QVMQHPYFSK IDWKNVRTAY RPPFVPDLNS EIDAGYFDDF 540
541 TNENDMSKYK EVHEKQAAIA NMVNTFNKPK RNAFIGFTFR HQKNSHPTSS SSALSSPLSA 600
601 PSFGTLL |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
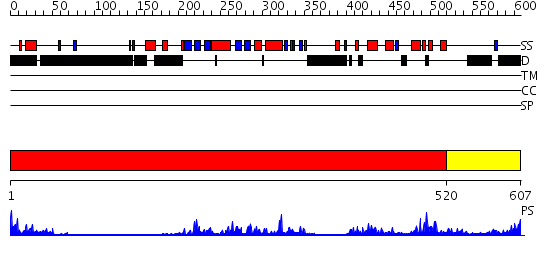
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..519] | 46.09691 | No description for 2fo0A was found. | |
| 2 | View Details | [520..607] | 53.09691 | Crystal Structure of ROCK 1 bound to fasudil |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)