













| General Information: |
|
| Name(s) found: |
cdtl-7 /
CE31401
[WormBase]
|
| Description(s) found:
Found 21 descriptions. SHOW ALL |
|
| Organism: | Caenorhabditis elegans |
| Length: | 730 amino acids |
Gene Ontology: |
|
| Cellular Component: |
cytoplasm
[IEA |
| Biological Process: |
locomotory behavior
[IMP signal transduction [IEA nematode larval development [IMP positive regulation of growth rate [IMP embryonic development ending in birth or egg hatching [IMP post-embryonic body morphogenesis [IMP protein amino acid phosphorylation [IEA positive regulation of multicellular organism growth [IMP growth [IMP |
| Molecular Function: |
magnesium ion binding
[IEA ATP binding [IEA protein tyrosine kinase activity [IEA non-membrane spanning protein tyrosine kinase activity [IEA protein kinase activity [IEA protein serine/threonine kinase activity [IEA |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MEISPGSSTH ERDRKGSYGH RERTRSHSGS PSRFYSKDKR GSSRQGVRPR DRDSKDSISP 60
61 QYKQRNWSRG GGGGGRDRGR NDFSYRKKGK DYNKRRDKRS RSRSRHRSPK RSGSSKKSKR 120
121 RNSSGSSSSD LMDTSLMSEL KKHGDYGSSS KSKKKSRKRR KHSSSSSSSS GEAMDLPVSS 180
181 NGMNVTAIPP PPSFNINPFQ PMFSQPPPPP LPPNSQFMTP PPRPPPAPFS IPPPSVDIHF 240
241 AATASFSLSS IPPPPPQTDG GASSSKRQDP LPMPPDSKRI ATRPVITTRR GHATNRPSDS 300
301 DSWYKTNLTH YTMLDQIGEG TYGQVYKAVN NLTGEQVALK RVRLENEKEG FPITAIREIK 360
361 ILRQLHHKNI VRLMDIVIDD ISMDELKRTR ANFYLVFEYV DHDLIGLLES KELVDFNKDQ 420
421 ICSLFKQLLE GLAYIHNTGF LHRDIKCSNI LVNNKGELKI ADLGLARLWE KESRLYTNRV 480
481 ITLWYRPPEL LLGDERYGPA IDVWSTGCML GELFTRKPLF NGNNEFGQLE LISKVCGSPN 540
541 VDNWPELTEL VGWNTFRMKR TYQRRIREEF EHIMPREAVD LLDKMLTLNP EKRISAKEAL 600
601 NHPWIRSLEH TTVQPLKLPQ HQDCHEMWSK KQKKSARLGR QAEGSSGSGH SIRATSHPRA 660
661 PTQPSTTTTK SNGSSNHHHH HHHSHHHASS LPPSGGHAPP PPPPPTQASS TSHNNHQPVP 720
721 QSQYQSVFFK |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
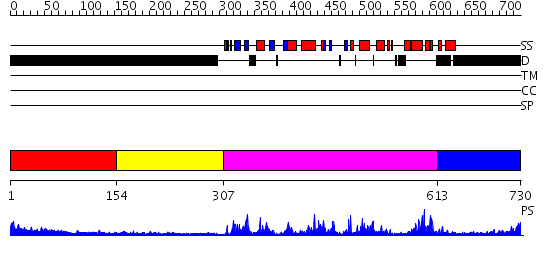
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..153] | 7.39794 | pak1 autoregulatory domain | |
| 2 | View Details | [154..306] | N/A | No confident structure predictions are available. | |
| 3 | View Details | [307..612] | 88.09691 | Structure of CDK2/Cyclin A with PNU-292137 | |
| 4 | View Details | [613..730] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)