













| General Information: |
|
| Name(s) found: |
jnk-1 /
CE27574
[WormBase]
|
| Description(s) found:
Found 23 descriptions. SHOW ALL |
|
| Organism: | Caenorhabditis elegans |
| Length: | 463 amino acids |
Gene Ontology: |
|
| Cellular Component: |
nucleus
[IDA cytoplasm [IDA neuronal cell body [IDA axon [IDA |
| Biological Process: |
protein amino acid phosphorylation
[IEA hyperosmotic response [IGI JUN phosphorylation [IDA |
| Molecular Function: |
ATP binding
[IEA protein tyrosine kinase activity [IEA JUN kinase activity [IDA MAP kinase activity [IEA protein kinase activity [IEA protein serine/threonine kinase activity [IEA |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MEERLSTTSS YPSHPGRSVE EDHNTLLASS SISSIIRGTR GHLNNFIESV GNWLVPSSSG 60
61 RDDDAVSLDS CQSVYSPVRH HINSGTGGGI LMEPSSIHVP ENYYSVTIGE AQMVVLKRYQ 120
121 NLRLIGSGAQ GIVCSAFDTV RNEQVAIKKL SRPFQNVTHA KRAYRELKLM SLVNHKNIIG 180
181 ILNCFTPQKK LDEFNDLYIV MELMDANLCQ VIQMDLDHER LSYLLYQMLC GIRHLHSAGI 240
241 IHRDLKPSNI VVRSDCTLKI LDFGLARTAI EAFMMTPYVV TRYYRAPEVI LGMGYKENVD 300
301 VWSIGCIFGE LIRGRVLFPG GDHIDQWTRI IEQLGTPDRS FLERLQPTVR NYVENRPRYQ 360
361 ATPFEVLFSD NMFPMTADSS RLTGAQARDL LSRMLVIDPE RRISVDDALR HPYVNVWFDE 420
421 IEVYAPPPLP YDHNMDVEQN VDSWREHIFR ELTDYARTHD IYS |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
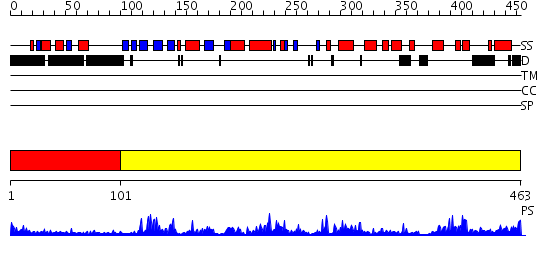
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..100] | 53.045757 | No description for 2fo0A was found. | |
| 2 | View Details | [101..463] | 96.69897 | Structural basis for the selective inhibition of JNK1 by the scaffolding protein JIP1 and SP600125 |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.98 |
Source: Reynolds et al. (2008)