













| General Information: |
|
| Name(s) found: |
akt-1 /
CE05274
[WormBase]
|
| Description(s) found:
Found 21 descriptions. SHOW ALL |
|
| Organism: | Caenorhabditis elegans |
| Length: | 546 amino acids |
Gene Ontology: |
|
| Cellular Component: |
intracellular
[IDA |
| Biological Process: |
dauer larval development
[IMP insulin receptor signaling pathway [IMP determination of adult lifespan [IMP protein amino acid phosphorylation [IEA |
| Molecular Function: |
ATP binding
[IEA protein tyrosine kinase activity [IEA zinc ion binding [IEA protein kinase activity [IEA protein serine/threonine kinase activity [IEA |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSMTSLSTKS RRQEDVVIEG WLHKKGEHIR NWRPRYFMIF NDGALLGFRA KPKEGQPFPE 60
61 PLNDFMIKDA ATMLFEKPRP NMFMVRCLQW TTVIERTFYA ESAEVRQRWI HAIESISKKY 120
121 KGTNANPQEE LMETNQQPKI DEDSEFAGAA HAIMGQPSSG HGDNCSIDFR ASMISIADTS 180
181 EAAKRDKITM EDFDFLKVLG KGTFGKVILC KEKRTQKLYA IKILKKDVII AREEVAHTLT 240
241 ENRVLQRCKH PFLTELKYSF QTNDRLCFVM EFAIGGDLYY HLNREVQMNK EGFSEPRARF 300
301 YGSEIVLALG YLHANSIVYR DLKLENLLLD KDGHIKIADF GLCKEEISFG DKTSTFCGTP 360
361 EYLAPEVLDD HDYGRCVDWW GVGVVMYEMM CGRLPFYSKD HNKLFELIMA GDLRFPSKLS 420
421 QEARTLLTGL LVKDPTQRLG GGPEDALEIC RADFFRTVDW EATYRKEIEP PYKPNVQSET 480
481 DTSYFDNEFT SQPVQLTPPS RSGALATVDE QEEMQSNFTQ FSFHNVMGSI NRIHEASEDN 540
541 EDYDMG |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
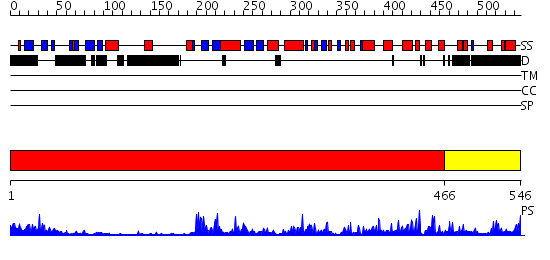
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..465] | 54.154902 | CHICKEN SRC TYROSINE KINASE | |
| 2 | View Details | [466..546] | 97.69897 | No description for 2jdoA was found. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.97 |
Source: Reynolds et al. (2008)