













| General Information: |
|
| Name(s) found: |
prp4 /
SPCC777.14
[Sanger Pombe]
|
| Description(s) found:
Found 21 descriptions. SHOW ALL |
|
| Organism: | Schizosaccharomyces pombe |
| Length: | 477 amino acids |
Gene Ontology: |
|
| Cellular Component: |
cellular_component
[ND]
|
| Biological Process: |
biological regulation
[RCA]
protein amino acid phosphorylation [TAS nuclear mRNA splicing, via spliceosome [TAS |
| Molecular Function: |
ATP binding
[ISS protein serine/threonine kinase activity [TAS |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSDDRFAEDE IIQQRRKRRL EILKKYQQTG NGHSDLSIPE KKLKEDVDQV STTKPIEAVP 60
61 KMKTNASKIE INKEGSNSNT KLDVTNSTTS DSPSIKSSVQ IEDTEDDMFA DSPSPSVKRQ 120
121 NTGKGISTLT RSFADMQDNW DDIEGYYKVV LMEELDSRYI VQSNLGKGMF STVVSALDRN 180
181 RNQTFAIKII RNNEVMYKEG LKEVSILERL QAADREGKQH IIHYERHFMH KNHLCMVFEM 240
241 LSLNLRDILK KFGRNVGLSI KAVRLYAYQM FMALDLLKQC NVIHSDIKPD NMLVNEKRNI 300
301 LKICDLGSAS DASENEITPY LVSRFYRAPE IILGFPYSCP IDTWSVGCSL YELYTGQILF 360
361 PGRTNNQMLR YMMECKGKFS HKMLKRSQFL NDHFDADFNF IQIDHDPITN QETRKPVKFS 420
421 KPTKDIRSRL KEVPTSTDEE FIIRQELMDL LEKCLELNPE KRVPPEVALK HPFFIKK |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
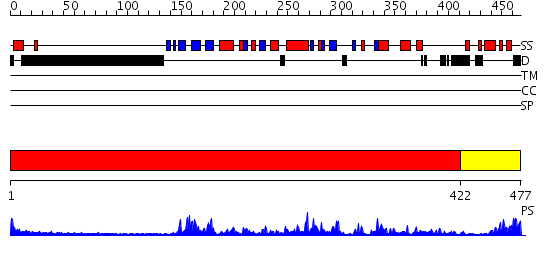
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..421] | 46.69897 | Abl tyrosine kinase, SH3 domain; Abl tyrosine kinase; Abelsone tyrosine kinase (abl) | |
| 2 | View Details | [422..477] | 52.0 | Crystal structure of human CLK1 in complex with 10Z-Hymenialdisine |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)