













| General Information: |
|
| Name(s) found: |
gi|62473051
[NCBI NR]
gi|28571918 [NCBI NR] gi|28381489 [NCBI NR] |
| Description(s) found:
Found 7 descriptions. SHOW ALL |
|
| Organism: | Drosophila melanogaster |
| Length: | 2063 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSNELSELIA LGCPDLAAQK PQGGSTQVEL ILNTERRLSI KQMTATDLAT LRAAAAAEAE 60
61 ARSRAQAEAE ALAIAQEQFR QAREAESKAR ARAALEVQAQ LQRVMESEKR RQQEEEERRI 120
121 RQQAEEEQED DEDDDNDEEE ELTKGSLPTN SGPRERIASL PCNIDDAMEM HLLNVSQPKA 180
181 SSKDDSPPKK STSHIADSIE LLRMQRAARD ARSHNRSRER SISPDRNRER SAAPDRLQSS 240
241 ASMSSLDSAS ASASRQPSKA GSRRGSTTSA TGVEPPLMAA PPQPTKFPLT KTVSAPNMDR 300
301 RRSSLTSLFP GPSPLVAPEP SLHTNPDPRV QEQIEEDRRR RRMDDGYREI PSGKMVHRTK 360
361 TPPPVGTNLG VSLKRVTAPS GSITIKPKES PMLGVVLRRV EKKTVPQKSI LDDDKPLYHF 420
421 SIVRSDHKEH VPAQKPKPKP APPAAKPSPG GILTGPQVIR NAPKQPVPVK PQRPMPGVPI 480
481 TITKIEGDKI IIIKKIIVPK NSKIPEQYLQ VGRRRVGDRL VVSVVRLVFG VSFLLCFVRN 540
541 LFYRIYLDAP SPSLRTKEES QPAMQKPTPP PASGQQFFQV VSPQFASVLS SSIPESKCAV 600
601 RSPISSPLAI RKSRPPLLPS SPKSTPPLPR KHHPLAYNAA TGTISSASGA ALPSRLMATI 660
661 ASSPASSISL SSCSSPSSSP PPPVPCRAPK NASRPVAVTA APPPSTSNEI SLDKLLQQQQ 720
721 ELEEKSAAAT NSAKMDANFY DAEIEMMNKY LKSLPDYSEL DRKLHQEFQE CEDLYDKIKR 780
781 QQQPLAKSNS QQSVTKAVGP GVPAISSGLS KSSSINFAQN PSSRGAAPRL AYPSIFSQAA 840
841 GEPKLQRSIS SSNMPISAPN SMRPLPTKNG LQSGSNLSLN KQLMNEFWSE NLTSSQKRQT 900
901 PKRTFWNYEK ICGAQLGDAG QPFKVDAKTA KKLAIFDPTV AEAAQKELQR PQQSQVPQNQ 960
961 PHKLQKNASL SHLDLKVRQA VTKDDLYKLI CNEQSPLAGS NFVSRVPVKQ QFPAQQQQVL1020
1021 PKSMSMTHVP GGGQPMGAPL LRTSSRTHIP SYMKNLPSLS RSTSNSAILM TQPRKEPPPK1080
1081 EPLKAPAPLP AIAAPTVGKP SGVLKSSSSS CVPSPRCAAA FFRRPQEANN PQQQHHQPLQ1140
1141 KPQQVAPIEE SGPGDSASLE RKSEKSNSTL STSSFTVTNC CTTHLPQLSK FTSSFHIAPT1200
1201 TATTTAATAT PTPTGAATTS DQQQQSGGTP AMVAANLEVP TSSSSSAATK RQKDVDNKLE1260
1261 KCLNDMLKLK TSSNNNSTSN SNNNAIMSHS LTGEHKDPKT ALEGPTSSSS SSSSKYIGES1320
1321 QIPVPVQLYD PQKPLLQQQQ QQQRICYPIG KSNSTSQLPM GGYQRLLQHQ QQQHHQQQQQ1380
1381 QHQEQQQYPQ HKRPFLNWNS FACSAMNGAS DPFMQQQHMP AHQQQQHLPH KLQQSYSSSH1440
1441 VPKQAPKSGL AMFLQKNTNK ENKFGQPMQQ QPPGMMPQMY GYQAPQQQSK IGYPRTGAPL1500
1501 THSASFSSAQ RPTALQFHQQ HQQQQHLQQQ QQHPQQQQHQ HSSFGVGMMS RNYYNMPKQP1560
1561 ERKPLQTFDP YAYPKPNQMQ PVKYQQQQQH PHTQFQNASA GGGGGGAAGL QYDPNTNTQL1620
1621 FYASPASSSS NKQPQQPQQQ QQQQQSQLQQ SNSVIFNHSG QQHQPHQQQQ NEMSKSALGL1680
1681 HFIETAKPVI QDDADGHLIY HTGDILHHRY KIMATLGEGT FGRVVKVKDM ERDYCMALKI1740
1741 IKNVEKYREA AKLEINALEK IAQKDPHCDH LCVKMIDWFD YHGHMCIVFE MLGLSVFDFL1800
1801 RENNYEPYPL DQVRHMAYQL CYSVKFLHDN RLTHTDLKPE NILFVDSDYT SHYNHKINRE1860
1861 VRRVKNTDVR LIDFGSATFD HEHHSTIVST RHYRAPEVIL ELGWSQPCDV WSIGCILFEL1920
1921 YLGITLFQTH DNREHLAMME RILGQIPYRM ARNHTLYSKT KTKYFYHGKL DWDEKSSAGR1980
1981 YVRDHCKPLF LCQLSDSEDH CELFSLIKKM LEYEPSSRIT LGEALHHPFF DRLPPHHRVG2040
2041 EVSNKQPLSS GSSSRERSHS LSR |
SHOWING SINGLE HITS. [ Hide Single Hits ]
New Feature: Upload Your Own Microscopy Data
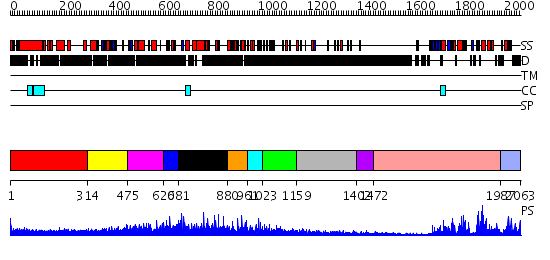
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..313] | 31.09691 | Heavy meromyosin subfragment | |
| 2 | View Details | [314..474] | 4.0 | No description for 2dm9A was found. | |
| 3 | View Details | [475..619] | 18.221849 | Solution structure of the first SH3 domain of human intersectin2 (KIAA1256) | |
| 4 | View Details | [620..680] | N/A | No confident structure predictions are available. | |
| 5 | View Details | [681..879] | N/A | No confident structure predictions are available. | |
| 6 | View Details | [880..960] | N/A | No confident structure predictions are available. | |
| 7 | View Details | [961..1022] | N/A | No confident structure predictions are available. | |
| 8 | View Details | [1023..1158] | 2.192994 | View MSA. No confident structure predictions are available. | |
| 9 | View Details | [1159..1401] | N/A | No confident structure predictions are available. | |
| 10 | View Details | [1402..1471] | N/A | No confident structure predictions are available. | |
| 11 | View Details | [1472..1986] | 57.154902 | Abl tyrosine kinase, SH3 domain; Abl tyrosine kinase; Abelsone tyrosine kinase (abl) | |
| 12 | View Details | [1987..2063] | 2.7 | Crystal structure of non-phosphorylated Fus3 |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 8 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 9 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 10 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 11 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 12 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.60 |
Source: Reynolds et al. (2008)