













| General Information: |
|
| Name(s) found: |
CG8908-PA /
FBpp0085588
[FlyBase]
|
| Description(s) found:
Found 12 descriptions. SHOW ALL |
|
| Organism: | Drosophila melanogaster |
| Length: | 1625 amino acids |
Gene Ontology: |
|
| Cellular Component: |
ATP-binding cassette (ABC) transporter complex
[ISS]
|
| Biological Process: |
transport
[ISS]
|
| Molecular Function: |
ATP binding
[IEA]
ATPase activity, coupled to transmembrane movement of substances [ISS] transporter activity [ISS] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSSGESPVKT QSSICLLWLV ICKTARFQFS NTLTSVIIIV GPIVVFLVYA ATALFREPQE 60
61 ETSVKYPPVN ITTKVPYIYY SPENLVLAAV IEDVVHDLKA VGSKAFASAS ELNKALIGKD 120
121 TYGYVGIEFD DSFSDINELP INVTVGLRFP LHLRKNPKMV WDNTSILKHS KMDVDYYQVE 180
181 GFLVVQAKLS EALIRAKNEA VVLPEVILQH YPDVAEVDRL DNRVALGGVL FLPVTISAAY 240
241 LAQMIVMERR DHLRDMLELM GVRAWIYWLS WFLVAFLLLS IPTVFMVLLL KWRYFSLSDS 300
301 SLVLFFLLVY NLEVLTSAFM ISSFFSDTVG VQVAIVIVHL IGCLPWRLLL MGYVPTLPRT 360
361 IFVCLFLNSS LAMGLQQFIK SENLRLGLHW TYLFERTDWD EFVHLGPILL FMLFGCLLRI 420
421 LVLAYMEQLR SYQNRKWNIE KVFNEWIAVK DLNLNFYQDE ITVFLGHNDS GKSTILMLLA 480
481 GFLRPSAGEI TINGYDLATN QRKARQSMCI CPQPNVLFEK VNARWHLQFY CRLKGLNRQE 540
541 ASAETDKYLE IGRLQDFANT KVKNLPSGIK RMLMLCCNLC GNSKILLLDE PGTSMDPAMR 600
601 SNMWDLLRRE RKGRCIIMAT HNMNEAEVVA DQIVVLCDAQ VIGYGTTGFL TQIAGTGSSY 660
661 LLICTKMDTC IVAEVTNFLQ IRFPDIKLHN EFSIYVTYEL PTKYVQQYAA LFLELEEALG 720
721 ELNLAEISVC APTLGSVFLQ MGEEMRQSWN RISSFDLLSS PSPMLSLLNL LPTFEVREDD 780
781 GRVKCCNQWR AIVEKKRLFT LRHQKFYCMI IATPIIICLL IISFSIFIFL VDHHLSELLV 840
841 TDLSIYPKAV FVIDSPAEGN RFERHYMANV IGQGGTVRST SGTPIDDYLL AEMESDQVKV 900
901 QHTFLAGATF DSNANSIIAW SNNKLKHGSA LSMGLVYAAI GQELAKLDIR IVNKPYQDTV 960
961 QQAVRGLIYA STIEFAVLVF HYLVLGTAIF AVLPIVERRS KVQHQQFSSG MSRSTYWLSH1020
1021 LSWDYCFYIA MILPLIVVAG ITIGSVLPVI VQLLAFGFSA ISFTYLLCLM SNDFGKMFSI1080
1081 ILYINMIGNY GFIVFLFGYC KETLFPGVLA LFIHPKSPQT RYAVIESVLL VHPHYSLCCG1140
1141 MYEAIRMSTF SLKQLPYLIF SGAAYLTIVV FAWVPRRLNY VFKLCLDQLC SSFMLTDYQL1200
1201 LFRSIRNEKI YPSYKDEDKE VDKMRRRLAY LTTAHYAFFP LILKNLSKRY GSFVAVRSLT1260
1261 LDLNPFECVG LLGRNGSGKS SIFRMIVGME SITVGSIHIK GYSLKTRPKD ASRHVGFCPR1320
1321 ELMLLSFMTG KDALRFCCLI NGIRREYIKS LVASLAECFE LVPHMNKRIS TYSNGTKRKL1380
1381 MIAMGTLAPS LMCLDEPTAG VDMHAKYEIW SILDGIRQGG RSILLTTHNL EECEFLCTNV1440
1441 GIMDHGSLLC YGSLSRLKHR FNMGIFVKVK MGTRAEMDDE RDNWMQITMM PPNDSEMGST1500
1501 VGARRMMLAH LLRHHNPPVE PVKKSRTSRA QPDTTNSELN MRKDYEALLQ ELEEVFKKDH1560
1561 PYSTVSEKYS YRGMITFCIP RGQIKWSAIF EYMENLKDEM QILYYSVSHT TFQDVFMKFV1620
1621 RKQNQ |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
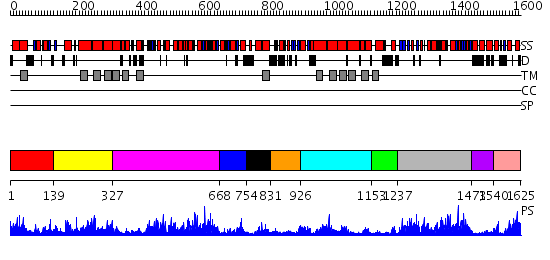
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..138] | 1.171989 | View MSA. No confident structure predictions are available. | |
| 2 | View Details | [139..326] | N/A | No confident structure predictions are available. | |
| 3 | View Details | [327..667] | 51.39794 | No description for 2hydA was found. | |
| 4 | View Details | [668..753] | 84.0 | Crystal Structure of ATPase subunit of ABC Sugar Transporter | |
| 5 | View Details | [754..830] | N/A | No confident structure predictions are available. | |
| 6 | View Details | [831..925] | N/A | No confident structure predictions are available. | |
| 7 | View Details | [926..1152] | N/A | No confident structure predictions are available. | |
| 8 | View Details | [1153..1236] | N/A | No confident structure predictions are available. | |
| 9 | View Details | [1237..1472] | 60.69897 | Crystal structure of ABC transporter ATP-binding protein (TM0544) from Thermotoga maritima at 2.10 A resolution | |
| 10 | View Details | [1473..1539] | 2.76 | Crystal structure of ABC transporter ATP-binding protein (TM0544) from Thermotoga maritima at 2.10 A resolution | |
| 11 | View Details | [1540..1625] | 1.246989 | View MSA. No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 8 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 9 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 10 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 11 | No functions predicted. |
| Source: Reynolds et al. 2008. Manuscript submitted |

|