













| General Information: |
|
| Name(s) found: |
AKT2
[HGNC (HUGO)]
|
| Description(s) found:
Found 35 descriptions. SHOW ALL |
|
| Organism: | Homo sapiens |
| Length: | 481 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MNEVSVIKEG WLHKRGEYIK TWRPRYFLLK SDGSFIGYKE RPEAPDQTLP PLNNFSVAEC 60
61 QLMKTERPRP NTFVIRCLQW TTVIERTFHV DSPDEREEWM RAIQMVANSL KQRAPGEDPM 120
121 DYKCGSPSDS STTEEMEVAV SKARAKVTMN DFDYLKLLGK GTFGKVILVR EKATGRYYAM 180
181 KILRKEVIIA KDEVAHTVTE SRVLQNTRHP FLTALKYAFQ THDRLCFVME YANGGELFFH 240
241 LSRERVFTEE RARFYGAEIV SALEYLHSRD VVYRDIKLEN LMLDKDGHIK ITDFGLCKEG 300
301 ISDGATMKTF CGTPEYLAPE VLEDNDYGRA VDWWGLGVVM YEMMCGRLPF YNQDHERLFE 360
361 LILMEEIRFP RTLSPEAKSL LAGLLKKDPK QRLGGGPSDA KEVMEHRFFL SINWQDVVQK 420
421 KLLPPFKPQV TSEVDTRYFD DEFTAQSITI TPPDRYDSLG LLELDQRTHF PQFSYSASIR 480
481 E |
SHOWING SINGLE HITS. [ Hide Single Hits ]
New Feature: Upload Your Own Microscopy Data
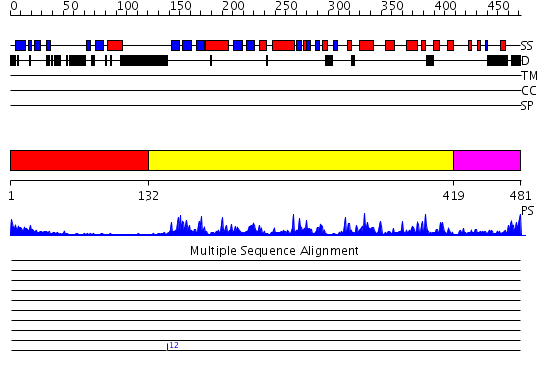
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..131] | 13.30103 | Solution Structure of the Pleckstrin Homology Domain of Human Protein Kinase B beta (Pkb/Akt) | |
| 2 | View Details | [132..418] | 62.045757 | No description for 2j4zA was found. | |
| 3 | View Details | [419..481] | 92.39794 | crystal structure of an inactive Akt2 kinase domain |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)