













| General Information: |
|
| Name(s) found: |
gi|30682811
[NCBI NR]
gi|30682806 [NCBI NR] gi|23308449 [NCBI NR] gi|22135803 [NCBI NR] |
| Description(s) found:
Found 13 descriptions. SHOW ALL |
|
| Organism: | Arabidopsis thaliana |
| Length: | 551 amino acids |
Gene Ontology: |
|
| Cellular Component: |
nucleus
[IDA]
cytoplasm [IDA] cytosol [IDA] plasma membrane [IDA] phragmoplast [IDA] |
| Biological Process: |
protein amino acid phosphorylation
[IEA][ISS]
|
| Molecular Function: |
ATP binding
[ISS]
protein kinase activity [ISS] kinase activity [ISS] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 METAKAWLSK LKSKDKVKSS KKKEATSNVK EGPKTAGGEE ALSNITKEKA AAAKLYIENH 60
61 YKMQMQSLQE RKERRKMLEK KLAAAEVSEE EQNNLLKDLE MKETEYMRRQ RHKMGADDFE 120
121 PLTMIGKGAF GEVRICREKG TGNVYAMKKL KKSEMLRRGQ VEHVKAERNL LAEVDSNCIV 180
181 KLYCSFQDEE YLYLIMEYLP GGDMMTLLMR KDTLTEDEAR FYIGETVLAI ESIHKHNYIH 240
241 RDIKPDNLLL DKDGHMKLSD FGLCKPLDCS NLQEKDFTVA RNVSGALQSD GRPVATRRTQ 300
301 QEQLLNWQRN RRMLAYSTVG TPDYIAPEVL LKKGYGMECD WWSLGAIMYE MLVGFPPFYS 360
361 DDPMTTCRKI VNWRNYLKFP DEVRLSPEAK DLICRLLCNV EQRLGTKGAD EIKGHPWFRG 420
421 TEWGKLYQMK AAFIPQVNDE LDTQNFEKFE ETDKQVPKSA KSGPWRKMLS SKDINFVGYT 480
481 YKNVEIVNDD QIPGIAELKK KSNKPKRPSI KSLFEDETSG GTTTHQGSFL NLLPTQIEDP 540
541 EKEGSKSSSS G |
SHOWING SINGLE HITS. [ Hide Single Hits ]
New Feature: Upload Your Own Microscopy Data
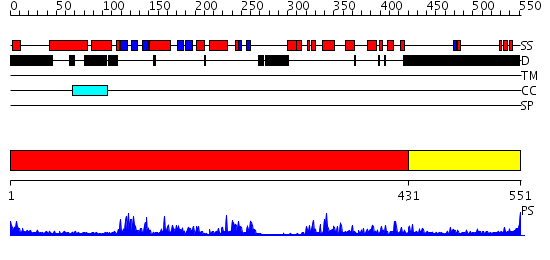
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..430] | 64.09691 | CRYSTAL STRUCTURE OF HUMAN TYROSINE-PROTEIN KINASE C-SRC | |
| 2 | View Details | [431..551] | 82.522879 | Crystal Structure of ROCK 1 bound to fasudil |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)