













| General Information: |
|
| Name(s) found: |
Y1461_ARATH
[Swiss-Prot]
|
| Description(s) found:
Found 17 descriptions. SHOW ALL |
|
| Organism: | Arabidopsis thaliana |
| Length: | 572 amino acids |
Gene Ontology: |
|
| Cellular Component: |
plasma membrane
[IDA]
|
| Biological Process: |
protein amino acid phosphorylation
[IEA]
|
| Molecular Function: |
ATP binding
[IEA]
protein tyrosine kinase activity [IEA] protein kinase activity [IEA] protein serine/threonine kinase activity [IEA] kinase activity [ISS] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MGCVFGREAA TTTTAEAKQA KSSKASSGVV VVGESSVTKS NGVIADDVEK KKNEEANGDK 60
61 ERKSSKGDRR RSTKPNPRLS NPSKHWRGEQ VAAGWPSWLS DACGEALNGW VPRKADTFEK 120
121 IDKIGQGTYS NVYKAKDMLT GKIVALKKVR FDNLEPESVK FMAREILVLR RLDHPNVVKL 180
181 EGLVTSRMSC SLYLVFQYMD HDLAGLASSP VVKFSESEVK CLMRQLISGL EHCHSRGVLH 240
241 RDIKGSNLLI DDGGVLKIAD FGLATIFDPN HKRPMTSRVV TLWYRAPELL LGATDYGVGI 300
301 DLWSAGCILA ELLAGRPIMP GRTEVEQLHK IYKLCGSPSE DYWKKGKFTH GAIYKPREPY 360
361 KRSIRETFKD FPPSSLPLID ALLSIEPEDR QTASAALKSE FFTSEPYACE PADLPKYPPS 420
421 KEIDAKRRDE ETRRQRAASK AQGDGARKNR HRDRSNRALP APEANAELQS NVDRRRLITH 480
481 ANAKSKSEKF PPPHQDGGAM GVPLGASQHI DPTFIPRDMV PSFTSSSFNF SKDEPPTQVQ 540
541 TWSGPLGHPI TGVSRKKKDN TKSSKGKRAV VA |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
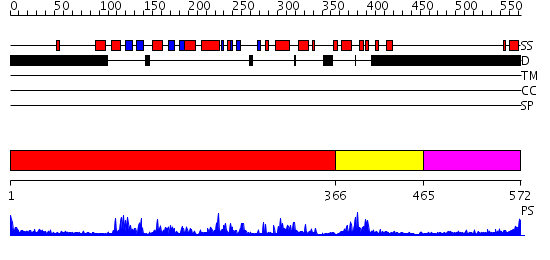
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..365] | 48.69897 | Carboxyl-terminal src kinase (csk) | |
| 2 | View Details | [366..464] | 2.36 | THE C-JUN N-TERMINAL KINASE (JNK3S) COMPLEXED WITH MGAMP-PNP | |
| 3 | View Details | [465..572] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.98 |
Source: Reynolds et al. (2008)