













| General Information: |
|
| Name(s) found: |
CG34120-PC /
FBpp0110124
[FlyBase]
|
| Description(s) found:
Found 16 descriptions. SHOW ALL |
|
| Organism: | Drosophila melanogaster |
| Length: | 1997 amino acids |
Gene Ontology: |
|
| Cellular Component: |
ATP-binding cassette (ABC) transporter complex
[ISS]
|
| Biological Process: |
transport
[ISS]
|
| Molecular Function: |
ATP binding
[IEA]
ATPase activity, coupled to transmembrane movement of substances [ISS] transporter activity [ISS] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MRVHLNGREV RELLRKDLLV RWRQKGLSLI LLAWPVMVFM LLYLIRLKYG SEELEACQYP 60
61 TRLLPTKNQV VPAAFSYICS IENRCQPAHP YEEYSHWKEA PLHSVIDVVN AFVTDERLHK 120
121 AVVELAEKAN FVSAITTLIT SDRFDIIRSN ISEIISLVPE IERRMNYSFD IKHLFSNRET 180
181 FVKLGVLLCG HPFPNTDTIP LVNEILESED FSQANDAEID AMPTRRRRRY EDLPTDWRPT 240
241 KYCKRLYRDV TSTNQGKLTW NTIKPIIQGR ILYTPANAMT ESVVKFSNAS FEELDRLKQL 300
301 SRAAATILTK LHTNATFQEA FDNLLKLAKS PLVKSLVGDD FDIGEIERVF EYIRTNQLIY 360
361 DILTTVADLM DCVSADRFEA VESVEELHKR AYELNQNKLF LAALNLEDVG LKQASYRLHM 420
421 DTDNTQPTFE NRNRFWFPGP ADSMVIDLKY HRGFVQLKQM VDLGIIKSKR EEAGFAPEED 480
481 GPESGRSLSG LFSIKQVEND TPDEDDDDFD LSLEESGDEK AAPKVSASAS DHEATTISPS 540
541 SLDGVTTEEV RVTTEQTVVS GDPDLLLLKN EDVLKRSKRQ GLFDLLGGFG GSGDANKKNK 600
601 FEVDNMQFYT KQFPYPAFLN DVFKRGLYLA QGVQVAYLLG LVVFVALSVR ERIWMRESRN 660
661 SMLMRSMGLK AHSELVAWAL MSFLELCVIF ALISGVLYSG GILGYTNWFF MMFYCLSFGL 720
721 CLISFCYMCT NFFNSANIGA VASALLFFIS LCPFIIVLMF DAKLSVFESF LVDLSFTTAF 780
781 AKGWGELIRM ELQQEGLTVR HLIQVGPARS ECAMALLMFL LDLLLYAVIG LAYQRYKKNN 840
841 YSFVKVSRSQ LDGKLGASLV NVSKLYGSKC AVSNLSLDFA RNQVSCLLGR NGAGKSTLIK 900
901 LLTGQIRQSS GKVLLAGEHQ VGVCWQDNIL IPTLTAREHL QLYAQIKIPP GGSGGVEEIR 960
961 SEVAQTLQSL NFGKHESYPS WQLSGGYRRR LCVAIAFIAS PSVVILDEPC NGVDAKARKD1020
1021 IWQLIERLRQ GRAVIFATHF MDEAKYLSDS LVIMRNGRII AQHSRDSLQR LCTSNYSIRL1080
1081 RCADATGVTF VIQKAQQLLP QTQVTHSGAA DYPHSLTINA SYAEHLTPGA VEFLELLQSQ1140
1141 VTAGSISDVE LTTSSSLEQE FEQLNRNGED AGPRRPASDR SGVVAGPAMI TDEPPTACKQ1200
1201 FRLLMGKRLR HLSRNYRLLL YVLLLPALFE LCAMWFVSYR LEDDFDTVLP LSRSLYPQTA1260
1261 QFLSHERATS FSEKLYPQLR TSCDHLGECR VFNTSQQSYD WVLNSWGEYS ERRYGGYGLN1320
1321 GSGATVWYNN KGYHSMMAWL NDLNSELLRT TMNDSESSIL TLNEPWKLGF AELSTSSILR1380
1381 QAGDGSMVFI LLIAFGLVVA SGSVYLVNER VNGEKLQQRL CGVSAVTYWL VALVWDYLVM1440
1441 VLGLIVCLVV ILMFGMPVFV DRQQLVGIIG LSLAFSFACV PSVHVAEKIF SDSSIAIVSI1500
1501 FCANLIVPLV TMGIILILGV VGDGPAWDDW RHALNQAFLI FPIHALGDGF LELCKNYMVA1560
1561 LVFRRYDIDS YKHPLASDLL GRHFTALLLV GVAALIINVL IEWHLLRRLW QRVERLLDCT1620
1621 YRRELDKLGQ LKLVNIQSIF KSCVDTGEAV RAENLWLAYR RGHYAVRNVN FSVQRGECFG1680
1681 LLGKNGAGKS TIFKLLTGQL QPDVGQIYFE QPGISYCPQS NPLDPLLTTT ECIRFYGRLR1740
1741 GIRDLDQFLD RVLDTYELRP YKDVQVRNLS GGNRRKLTVA VTCCGCTPTV LMDEPTSDMD1800
1801 PVTRDMVYAT IEQLLLARRA VVLTSHSVSE IEHLCQRVAV LRAGQVIASD SPQRLKSEHG1860
1861 GYYAVTCFCG PAQQAILSRS LNQRLPGARD LQHYAHSLRF LVRVRSPGSL GDAPLLSELF1920
1921 AILCDVCVNV ARFSLSRCRF ETVFERILDS SESNGSNGVH KDQQQQVAKN DLPSKSPAVG1980
1981 GTLETGYIHC GYEETRT |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
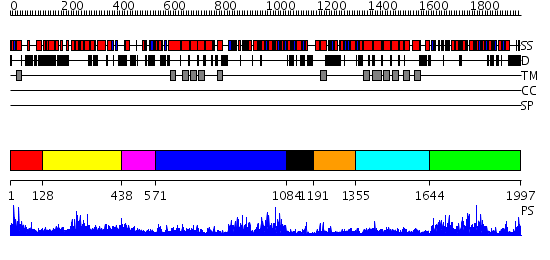
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..127] | 1.033993 | View MSA. No confident structure predictions are available. | |
| 2 | View Details | [128..437] | N/A | No confident structure predictions are available. | |
| 3 | View Details | [438..570] | N/A | No confident structure predictions are available. | |
| 4 | View Details | [571..1083] | 66.221849 | No description for 2hydA was found. | |
| 5 | View Details | [1084..1190] | 68.39794 | Glucose transport protein GlcV, N-terminal domain | |
| 6 | View Details | [1191..1354] | N/A | No confident structure predictions are available. | |
| 7 | View Details | [1355..1643] | N/A | No confident structure predictions are available. | |
| 8 | View Details | [1644..1997] | 85.0 | No description for 2d62A was found. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 8 | No functions predicted. |
| Source: Reynolds et al. 2008. Manuscript submitted |

|