













| General Information: |
|
| Name(s) found: |
IME2 /
YJL106W
[SGD]
|
| Description(s) found:
Found 28 descriptions. SHOW ALL |
|
| Organism: | Saccharomyces cerevisiae |
| Length: | 645 amino acids |
Gene Ontology: |
|
| Cellular Component: |
nucleus
[IDA |
| Biological Process: |
regulation of meiosis
[IMP protein amino acid phosphorylation [TAS |
| Molecular Function: |
protein kinase activity
[IDA |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MVEKRSRQSS SSGSEFSVPP DVDNPPLSIP LKTLSDRYQL IEKLGAGSFG CVTLAKAQFP 60
61 LSNILGKQHD IRGTLMDQPK NGHQNYITKT QGVVAIKTMM TKLHTLQDYT RVREIKFILA 120
121 IPANDHLIQI FEVFIDSENY QLHIVMECME QNLYQMMKHR RRRVFSIPSL KSILSQILAG 180
181 LKHIHEHNFF HRDLKPENIL ITPSTQYFEK EYMNQIGYQD NYVIKLADFG LARHVENKNP 240
241 YTAYVSTRWY RSPEILLRSG YYSKPLDIWA FGCVAVEVTV FRALFPGANE IDQIWKILEV 300
301 LGTPIKRSDF VNTNHITAPP PGGFWDDASN LVHKLNLKLP YVEGSSLDHL LSSSQLSDLS 360
361 EVVKKCLRWD PNERATAQEL CEMPFFENTV ASQVDARGNV TNTEQALIFA GINPVATNTK 420
421 PIYFNSSTKL PAETESNDID ISNNDHDSHA MCSPTLNQEK LTLVEFLNEF VEEDNDDHSI 480
481 PDVGTDSTIS DSIDETELSK EIRNNLALCQ LPDEEVLDHS LSNIRQLTND IEIINKDEAD 540
541 NMEQLFFDLE IPEKDEFQRK QPFNEHADID EDIVLPYVNN SNYTHTDRSH HRGDNVLGDA 600
601 SLGDSFNSMP DFTPRNFLIP TLKKSREKFE PHLSNSNQHF GNVTF |
| PUBLICATION | TOPOLOGY | COCOMPLEXED PROTEINS | |
| View Details | Ho Y, et al. (2002) |
|
CCT2 IME2 TCP1 |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
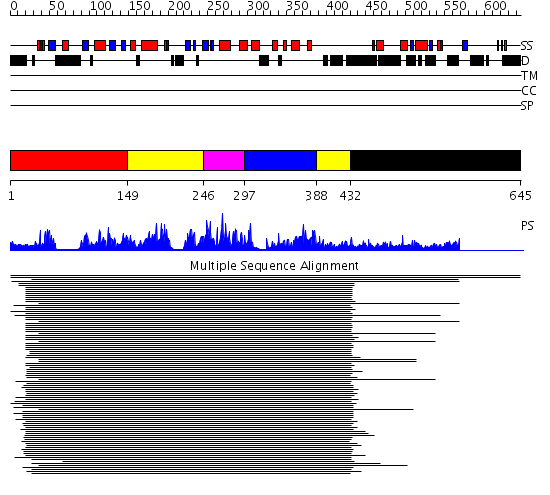
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..148] | 472.218487 | Cyclin-dependent PK, CDK2 | |
| 2 | View Details | [149..245] [388..431] |
472.218487 | Cyclin-dependent PK, CDK2 | |
| 3 | View Details | [246..296] | 472.218487 | Cyclin-dependent PK, CDK2 | |
| 4 | View Details | [297..387] | 472.218487 | Cyclin-dependent PK, CDK2 | |
| 5 | View Details | [432..645] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.90 |
Source: Reynolds et al. (2008)