













| General Information: |
|
| Name(s) found: |
TOR_ARATH
[Swiss-Prot]
|
| Description(s) found:
Found 9 descriptions. SHOW ALL |
|
| Organism: | Arabidopsis thaliana |
| Length: | 2481 amino acids |
Gene Ontology: |
|
| Cellular Component: |
chloroplast
[IEA]
|
| Biological Process: |
embryonic development ending in seed dormancy
[IMP]
|
| Molecular Function: |
protein binding
[IPI]
1-phosphatidylinositol-3-kinase activity [ISS] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSTSSQSFVA GRPASMASPS QSHRFCGPSA TASGGGSFDT LNRVIADLCS RGNPKEGAPL 60
61 AFRKHVEEAV RDLSGEASSR FMEQLYDRIA NLIESTDVAE NMGALRAIDE LTEIGFGENA 120
121 TKVSRFAGYM RTVFELKRDP EILVLASRVL GHLARAGGAM TSDEVEFQMK TAFDWLRVDR 180
181 VEYRRFAAVL ILKEMAENAS TVFNVHVPEF VDAIWVALRD PQLQVRERAV EALRACLRVI 240
241 EKRETRWRVQ WYYRMFEATQ DGLGRNAPVH SIHGSLLAVG ELLRNTGEFM MSRYREVAEI 300
301 VLRYLEHRDR LVRLSITSLL PRIAHFLRDR FVTNYLTICM NHILTVLRIP AERASGFIAL 360
361 GEMAGALDGE LIHYLPTIMS HLRDAIAPRK GRPLLEAVAC VGNIAKAMGS TVETHVRDLL 420
421 DVMFSSSLSS TLVDALDQIT ISIPSLLPTV QDRLLDCISL VLSKSHYSQA KPPVTIVRGS 480
481 TVGMAPQSSD PSCSAQVQLA LQTLARFNFK GHDLLEFARE SVVVYLDDED AATRKDAALC 540
541 CCRLIANSLS GITQFGSSRS TRAGGRRRRL VEEIVEKLLR TAVADADVTV RKSIFVALFG 600
601 NQCFDDYLAQ ADSLTAIFAS LNDEDLDVRE YAISVAGRLS EKNPAYVLPA LRRHLIQLLT 660
661 YLELSADNKC REESAKLLGC LVRNCERLIL PYVAPVQKAL VARLSEGTGV NANNNIVTGV 720
721 LVTVGDLARV GGLAMRQYIP ELMPLIVEAL MDGAAVAKRE VAVSTLGQVV QSTGYVVTPY 780
781 KEYPLLLGLL LKLLKGDLVW STRREVLKVL GIMGALDPHV HKRNQQSLSG SHGEVPRGTG 840
841 DSGQPIPSID ELPVELRPSF ATSEDYYSTV AINSLMRILR DASLLSYHKR VVRSLMIIFK 900
901 SMGLGCVPYL PKVLPELFHT VRTSDENLKD FITWGLGTLV SIVRQHIRKY LPELLSLVSE 960
961 LWSSFTLPGP IRPSRGLPVL HLLEHLCLAL NDEFRTYLPV ILPCFIQVLG DAERFNDYTY1020
1021 VPDILHTLEV FGGTLDEHMH LLLPALIRLF KVDAPVAIRR DAIKTLTRVI PCVQVTGHIS1080
1081 ALVHHLKLVL DGKNDELRKD AVDALCCLAH ALGEDFTIFI ESIHKLLLKH RLRHKEFEEI1140
1141 HARWRRREPL IVATTATQQL SRRLPVEVIR DPVIENEIDP FEEGTDRNHQ VNDGRLRTAG1200
1201 EASQRSTKED WEEWMRHFSI ELLKESPSPA LRTCAKLAQL QPFVGRELFA AGFVSCWAQL1260
1261 NESSQKQLVR SLEMAFSSPN IPPEILATLL NLAEFMEHDE KPLPIDIRLL GALAEKCRVF1320
1321 AKALHYKEME FEGPRSKRMD ANPVAVVEAL IHINNQLHQH EAAVGILTYA QQHLDVQLKE1380
1381 SWYEKLQRWD DALKAYTLKA SQTTNPHLVL EATLGQMRCL AALARWEELN NLCKEYWSPA1440
1441 EPSARLEMAP MAAQAAWNMG EWDQMAEYVS RLDDGDETKL RGLASPVSSG DGSSNGTFFR1500
1501 AVLLVRRAKY DEAREYVERA RKCLATELAA LVLESYERAY SNMVRVQQLS ELEEVIEYYT1560
1561 LPVGNTIAEE RRALIRNMWT QRIQGSKRNV EVWQALLAVR ALVLPPTEDV ETWLKFASLC1620
1621 RKSGRISQAK STLLKLLPFD PEVSPENMQY HGPPQVMLGY LKYQWSLGEE RKRKEAFTKL1680
1681 QILTRELSSV PHSQSDILAS MVSSKGANVP LLARVNLKLG TWQWALSSGL NDGSIQEIRD1740
1741 AFDKSTCYAP KWAKAWHTWA LFNTAVMSHY ISRGQIASQY VVSAVTGYFY SIACAANAKG1800
1801 VDDSLQDILR LLTLWFNHGA TADVQTALKT GFSHVNINTW LVVLPQIIAR IHSNNRAVRE1860
1861 LIQSLLIRIG ENHPQALMYP LLVACKSISN LRRAAAQEVV DKVRQHSGAL VDQAQLVSHE1920
1921 LIRVAILWHE MWHEALEEAS RLYFGEHNIE GMLKVLEPLH DMLDEGVKKD STTIQERAFI1980
1981 EAYRHELKEA HECCCNYKIT GKDAELTQAW DLYYHVFKRI DKQLASLTTL DLESVSPELL2040
2041 LCRDLELAVP GTYRADAPVV TISSFSRQLV VITSKQRPRK LTIHGNDGED YAFLLKGHED2100
2101 LRQDERVMQL FGLVNTLLEN SRKTAEKDLS IQRYSVIPLS PNSGLIGWVP NCDTLHHLIR2160
2161 EHRDARKIIL NQENKHMLSF APDYDNLPLI AKVEVFEYAL ENTEGNDLSR VLWLKSRSSE2220
2221 VWLERRTNYT RSLAVMSMVG YILGLGDRHP SNLMLHRYSG KILHIDFGDC FEASMNREKF2280
2281 PEKVPFRLTR MLVKAMEVSG IEGNFRSTCE NVMQVLRTNK DSVMAMMEAF VHDPLINWRL2340
2341 FNFNEVPQLA LLGNNNPNAP ADVEPDEEDE DPADIDLPQP QRSTREKEIL QAVNMLGDAN2400
2401 EVLNERAVVV MARMSHKLTG RDFSSSAIPS NPIADHNNLL GGDSHEVEHG LSVKVQVQKL2460
2461 INQATSHENL CQNYVGWCPF W |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
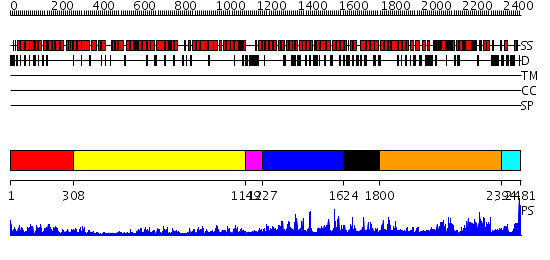
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..307] | 35.045757 | Importin beta | |
| 2 | View Details | [308..1148] | 67.69897 | No description for 2h4mA was found. | |
| 3 | View Details | [1149..1226] | 72.221849 | No description for 2iaeA was found. | |
| 4 | View Details | [1227..1623] | 2.05 | The superhelical TPR domain of O-linked GlcNAc transferase reveals structural similarities to importin alpha. | |
| 5 | View Details | [1624..1799] | 7.69897 | Crystal structure of an 8 repeat consensus TPR superhelix (trigonal crystal form) | |
| 6 | View Details | [1800..2393] | 94.69897 | Phoshoinositide 3-kinase (PI3K) helical domain; Phoshoinositide 3-kinase (PI3K); Phoshoinositide 3-kinase (PI3K), catalytic domain | |
| 7 | View Details | [2394..2481] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.52 |
Source: Reynolds et al. (2008)