













| General Information: |
|
| Name(s) found: |
mpk-2 /
CE36972
[WormBase]
|
| Description(s) found:
Found 15 descriptions. SHOW ALL |
|
| Organism: | Caenorhabditis elegans |
| Length: | 570 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: |
protein amino acid phosphorylation
[IEA |
| Molecular Function: |
magnesium ion binding
[IEA ATP binding [IEA protein tyrosine kinase activity [IEA MAP kinase activity [IEA protein kinase activity [IEA protein serine/threonine kinase activity [IEA |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MAAPAESHAR LDGRFWLEGT PYLAEENVGA GAYGVVCKAM DTRNKKQVAI KKIPRAFTAH 60
61 TLAKRSLREV RILRELLHEN IIAVLDMFTA EGAHGKDIYL VMDLMETDLH QILHSRQTLM 120
121 EQHFQYFFYQ LLRGLKYLHS AGIIHRDLKP SNLLLNGDCL LRIADFGMAR AYASASTVRD 180
181 DANVGGHMTQ YVSTRWYRAP EILFSMVEYD TKVDLWSAGC IFAEMLLRRQ LFPGKDSVSQ 240
241 IKMIVYYLGS PEEEVINRIT SDLVRDSIEA CGRKTPLPFS AIFPKASPEA RNMVSYLLQI 300
301 SPWKRYSADQ ILQHPFMAQY HNDQYEPLCP PRVQVDVDAI ERFEGAQVVA GLDEEARIFE 360
361 MRRGTNYDTR KTPPPYLDDK SNQTFEPDKE GKMGPREDPT DYLGVIRQFQ KTREQKDDDI 420
421 SDSDETPTSL SSAETIDTVR EVELHRKCSE ENYSSDSGME PSTSNANLIV QEIFKNGINW 480
481 PLDVTSISSA PPMLSGGIPP SKKSKNHLDR RNIDRIREGY SEKRRFIPKI RDKRASSDSS 540
541 SKLDDKDRIV RLRKSRQELR KTSKDDDRGV |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
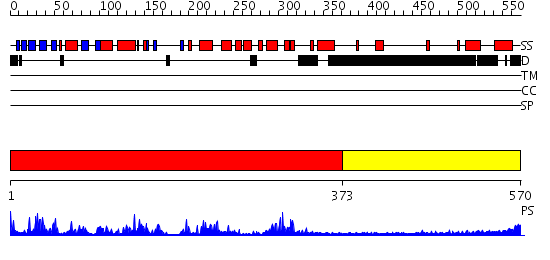
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..372] | 90.69897 | No description for 2gtmA was found. | |
| 2 | View Details | [373..570] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)