













| General Information: |
|
| Name(s) found: |
CE29020
[WormBase]
|
| Description(s) found:
Found 20 descriptions. SHOW ALL |
|
| Organism: | Caenorhabditis elegans |
| Length: | 470 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: |
nematode larval development
[IMP embryonic development ending in birth or egg hatching [IMP protein amino acid phosphorylation [IEA growth [IMP |
| Molecular Function: |
magnesium ion binding
[IEA ATP binding [IEA protein tyrosine kinase activity [IEA MAP kinase activity [IEA protein kinase activity [IEA protein serine/threonine kinase activity [IEA |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MTDDVDTHIH EKFDLQKRLG KGAYGIVWKA YDKRSRETVA LKKIFDAFRN PTDSQRTFRE 60
61 VMFLQEFGKH PNVIKLYNIF RADNDRDIYL AFEFMEADLH NVIKKGSILK DVHKQYIMCQ 120
121 LFRAIRFLHS GNVLHRDLKP SNVLLDADCR VKLADFGLAR SLSSLEDYPE GQKMPDLTEY 180
181 VATRWYRSPE ILLAAKRYTK GVDMWSLGCI LAEMLIGRAL FPGSSTINQI ERIMNTIAKP 240
241 SRADIASIGS HYAASVLEKM PQRPRKPLDL IITQSQTAAI DMVQRLLIFA PQKRLTVEQC 300
301 LVHPYVVQFH NPSEEPVLNY EVYPPLPDHI QLSIDDYRDR LYEMIDEKKA SFKRIQHEKI 360
361 RPYGEDKSRA PIAQAECSDT DYDTARSLQR TTSMDKNNSS SHDSSSGTLR ERAASAESRT 420
421 SKDSNGEMRN GNGNTPSSIK QRRRSVERAR LFANIKPSKI LHPHKLISNY |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
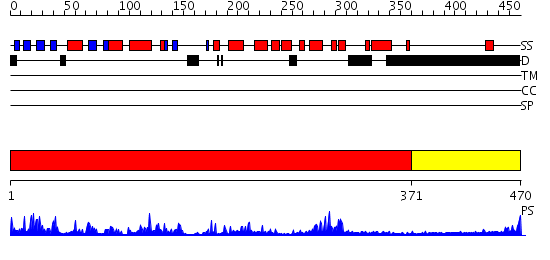
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..370] | 86.221849 | No description for 2gtmA was found. | |
| 2 | View Details | [371..470] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.82 |
Source: Reynolds et al. (2008)