













| General Information: |
|
| Name(s) found: |
SPCC550.03c
[Sanger Pombe]
|
| Description(s) found:
Found 17 descriptions. SHOW ALL |
|
| Organism: | Schizosaccharomyces pombe |
| Length: | 1213 amino acids |
Gene Ontology: |
|
| Cellular Component: |
cytoplasm
[ISS Ski complex [ISS] cytosol [IDA nucleolus [ISS |
| Biological Process: |
regulation of translation
[ISS mRNA catabolic process [ISS |
| Molecular Function: |
ATP binding
[IEA]
RNA helicase activity [ISS ATP-dependent helicase activity [IEA] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSSKLVDAIN EVAVSNKDKI ELDGIKDDSF DGHLSITEAT YDVDTFLKPS PALSKDWIRK 60
61 LQKKWDREIT YKGLYEYPET LARTQIRFQR HGLEGKIMGY KEVPELIEDL NSKNSSSFLR 120
121 KPSSKNEFVR GSTSNIPFLA DDSDVDAIAG EPSVKMALYG EDGLLQVPPG FSRGLSMTAT 180
181 STTDNLNDEF DPEKWDTKKV KSSNRNFVTI HELNEHLKNV NSKHSEIDDL LPDKRSIVSL 240
241 PPSTLNLHKQ PDYAHVVDSS APIENFQQLV PEMALDFPFE LDNFQKEAIY HLEMGDSVFV 300
301 AAHTSAGKTV VAEYAIALAQ KHMTKAIYTS PIKALSNQKF RDFKHKFEDV GILTGDVQVN 360
361 PEGSCLLMTT EILRSMLYRG ADLIRDVEFV IFDEVHYVND LERGVVWEEV IIMLPPHVTL 420
421 ILLSATVPNT KEFASWVGRT KKKNIYVIST LKRPVPLEHY LWVKQNMFKI VDQHGRFLMD 480
481 GYKSANDALK KPDKPVIAKD NKNSARGRGA ARGRGVQTNM MRGRGSAKSV ERRDANTWVH 540
541 LIGHLHKQNL LPVIVFVFSK KRCEEYVDTL TNRDLNNHQE KSEVHVVIEK AVARLKKEDR 600
601 LLPQIGRMRE MLSRGLAVHH GGLLPIIKEI VEILFQRGLV KVLFATETFA MGVNMPAKSV 660
661 VFSGTQKHDG RNFRDLLPGE YTQCSGRAGR RGLDVTGTVI ILSRSELPDT ASLRHMIMGP 720
721 SSKLISQFRL TYNMILNLLR VETLRIEDMI KRSFSENVNQ TLVPQHEEKI KSFEEKLSAL 780
781 KKEMSDVDLK EIKSCLLSSE SFKEYTKKMH FRAITTANGK RIFKDGRVIV FQQLDFTRTV 840
841 GVLLGTSIRT NASDCTLEVA YLNPQNNLKR PSDLLAFADA FNDVYDNAIF DESNQFKYGL 900
901 INLSGIERVC NTILRIDSGG IRDRRGGAFR KLSEQFASIK KFSDLLFEEV NWSKVRDFEF 960
961 CEAFEKRNFL QNKLSGNPII STPNFLTHFA LAYQEYELES NIDNLSSYIS DQNLELLPDY1020
1021 EQRIKVLQEL GYIDAERTVL LKGRVACEIN STSELVLTEL ILENSLADFS CEETIALLSA1080
1081 FVFDEKTEVE PTISPHLQKG KEMILSVAEK VNQIQEHYQV LYFNEGNDFE SQPRFGLMEV1140
1141 CYEWARGMSF NRITDLTDVL EGSIVRTIIR LDEVLRECRG AARVVGDSSM YTKMEECQNL1200
1201 IRRNIVFCPS LYM |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
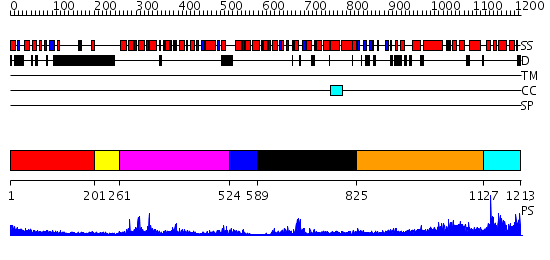
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..200] | N/A | No confident structure predictions are available. | |
| 2 | View Details | [201..260] | 60.69897 | No description for 2db3A was found. | |
| 3 | View Details | [261..523] | 23.0 | No description for 2p6rA was found. | |
| 4 | View Details | [524..588] | 16.0 | No description for 2p6nA was found. | |
| 5 | View Details | [589..824] | 7.0 | Nucleotide excision repair enzyme UvrB | |
| 6 | View Details | [825..1126] | 32.69897 | No description for 2q0zX was found. | |
| 7 | View Details | [1127..1213] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)