













| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSSILPFTPP IVKRLLGWKK GEQNGQEEKW CEKAVKSLVK KLKKTGQLDE LEKAITTQNV 60
61 NTKCITIPRS LDGRLQVSHR KGLPHVIYCR LWRWPDLHSH HELRAMELCE FAFNMKKDEV 120
121 CVNPYHYQRV ETPVLPPVLV PRHTEIPAEF PPLDDYSHSI PENTNFPAGI EPQSNIPETP 180
181 PPGYLSEDGE TSDHQMNHSM DAGSPNLSPN PMSPAHNNLD LQPVTYCEPA FWCSISYYEL 240
241 NQRVGETFHA SQPSMTVDGF TDPSNSERFC LGLLSNVNRN AAVELTRRHI GRGVRLYYIG 300
301 GEVFAECLSD SAIFVQSPNC NQRYGWHPAT VCKIPPGCNL KIFNNQEFAA LLAQSVNQGF 360
361 EAVYQLTRMC TIRMSFVKGW GAEYRRQTVT STPCWIELHL NGPLQWLDKV LTQMGSPSIR 420
421 CSSVS |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
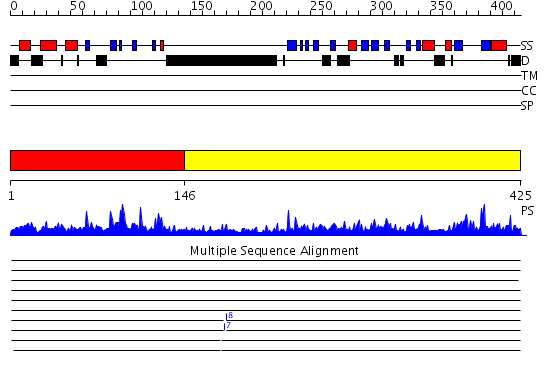
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..145] | 62.522879 | Crystal structure of Smad3-MH1 bound to DNA at 2.4 A resolution | |
| 2 | View Details | [146..425] | 103.0 | Smad2 MH2 domain |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)