













| General Information: |
|
| Name(s) found: |
STE11_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 717 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MEQTQTAEGT DLLIGDEKTN DLPFVQLFLE EIGCTQYLDS FIQCNLVTEE EIKYLDKDIL 60
61 IALGVNKIGD RLKILRKSKS FQRDKRIEQV NRLKNLMEKV SSLSTATLSM NSELIPEKHC 120
121 VIFILNDGSA KKVNVNGCFN ADSIKKRLIR RLPHELLATN SNGEVTKMVQ DYDVFVLDYT 180
181 KNVLHLLYDV ELVTICHAND RVEKNRLIFV SKDQTPSDKA ISTSKKLYLR TLSALSQVGP 240
241 SSSNLLAQNK GISHNNAEGK LRIDNTEKDR IRQIFNQRPP SEFISTNLAG YFPHTDMKRL 300
301 QKTMRESFRH SARLSIAQRR PLSAESNNIG DILLKHSNAV DMALLQGLDQ TRLSSKLDTT 360
361 KIPKLAHKRP EDNDAISNQL ELLSVESGEE EDHDFFGEDS DIVSLPTKIA TPKNWLKGAC 420
421 IGSGSFGSVY LGMNAHTGEL MAVKQVEIKN NNIGVPTDNN KQANSDENNE QEEQQEKIED 480
481 VGAVSHPKTN QNIHRKMVDA LQHEMNLLKE LHHENIVTYY GASQEGGNLN IFLEYVPGGS 540
541 VSSMLNNYGP FEESLITNFT RQILIGVAYL HKKNIIHRDI KGANILIDIK GCVKITDFGI 600
601 SKKLSPLNKK QNKRASLQGS VFWMSPEVVK QTATTAKADI WSTGCVVIEM FTGKHPFPDF 660
661 SQMQAIFKIG TNTTPEIPSW ATSEGKNFLR KAFELDYQYR PSALELLQHP WLDAHII |
SHOWING SINGLE HITS. [ Hide Single Hits ]
New Feature: Upload Your Own Microscopy Data
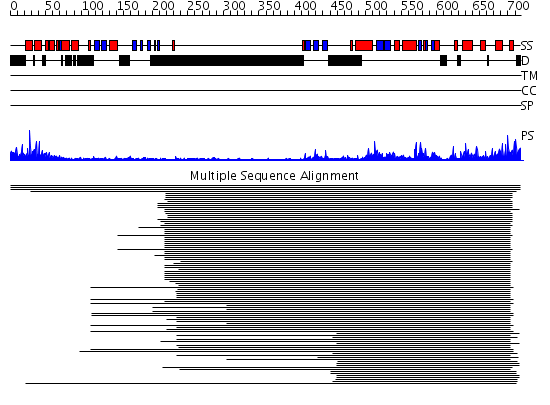
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..106] | 8.30103 | NMR structure of the Saccharomyces cerevisiae SAM (Sterile Alpha Motif) domain | |
| 2 | View Details | [107..210] | 2.33 | Protein kinase byr2 | |
| 3 | View Details | [211..717] | 66.39794 | No description for 2h8hA was found. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)