













| General Information: |
|
| Name(s) found: |
SLT2_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 484 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MADKIERHTF KVFNQDFSVD KRFQLIKEIG HGAYGIVCSA RFAEAAEDTT VAIKKVTNVF 60
61 SKTLLCKRSL RELKLLRHFR GHKNITCLYD MDIVFYPDGS INGLYLYEEL MECDMHQIIK 120
121 SGQPLTDAHY QSFTYQILCG LKYIHSADVL HRDLKPGNLL VNADCQLKIC DFGLARGYSE 180
181 NPVENSQFLT EYVATRWYRA PEIMLSYQGY TKAIDVWSAG CILAEFLGGK PIFKGKDYVN 240
241 QLNQILQVLG TPPDETLRRI GSKNVQDYIH QLGFIPKVPF VNLYPNANSQ ALDLLEQMLA 300
301 FDPQKRITVD EALEHPYLSI WHDPADEPVC SEKFEFSFES VNDMEDLKQM VIQEVQDFRL 360
361 FVRQPLLEEQ RQLQLQQQQQ QQQQQQQQQQ QPSDVDNGNA AASEENYPKQ MATSNSVAPQ 420
421 QESFGIHSQN LPRHDADFPP RPQESMMEMR PATGNTADIP PQNDNGTLLD LEKELEFGLD 480
481 RKYF |
SHOWING SINGLE HITS. [ Hide Single Hits ]
New Feature: Upload Your Own Microscopy Data
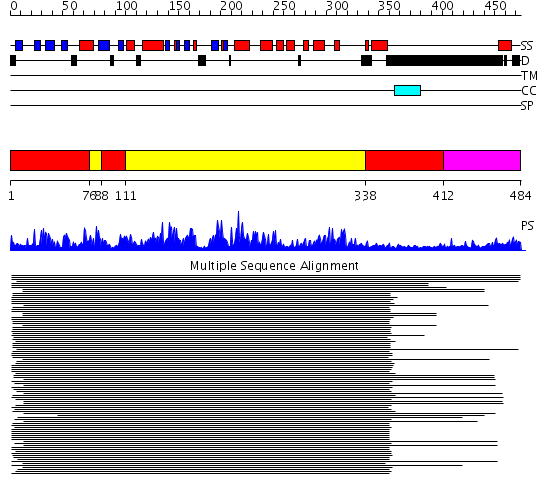
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..75] [88..110] [338..411] |
1008.54902 | MAP kinase Erk2 | |
| 2 | View Details | [76..87] [111..337] |
1008.54902 | MAP kinase Erk2 | |
| 3 | View Details | [412..484] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)