













| General Information: |
|
| Name(s) found: |
KDX1_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 433 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MATDTERCIF RAFGQDFILN KHFHLTGKIG RGSHSLICSS TYTESNEETH VAIRKIPNAF 60
61 GNKLSCKRTL RELKLLRHLR GHPNIVWLFD TDIVFYPNGA LNGVYLYEEL MECDLSQIIR 120
121 SEQRLEDAHF QSFIYQILCA LKYIHSANVL HCDLKPKNLL VNSDCQLKIC NFGLSCSYSE 180
181 NHKVNDGFIK GYITSIWYKA PEILLNYQEC TKAVDIWSTG CILAELLGRK PMFEGKDYVD 240
241 HLNHILQILG TPPEETLQEI ASQKVYNYIF QFGNIPGRSF ESILPGANPE ALELLKKMLE 300
301 FDPKKRITVE DALEHPYLSM WHDIDEEFSC QKTFRFEFEH IESMAELGNE VIKEVFDFRK 360
361 VVRKHPISGD SPSSSLSLED AIPQEVVQVH PSRKVLPSYS PEFSYVSQLP SLTTTQPYQN 420
421 LMGISSNSFQ GVN |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
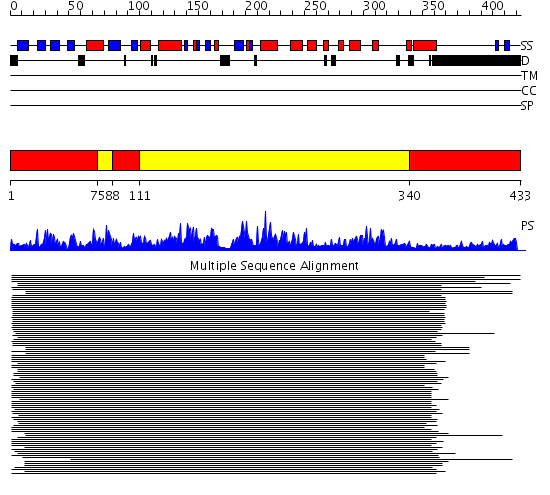
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..74] [88..110] [340..433] |
858.0103 | MAP kinase Erk2 | |
| 2 | View Details | [75..87] [111..339] |
858.0103 | MAP kinase Erk2 |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)