













| General Information: |
|
| Name(s) found: |
gi|207343070
[NCBI NR]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae AWRI1631 |
| Length: | 435 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MTTNEEFIRT QIFGTVFEIT NRYNDLNPVG MGAFGLVCSA TDTLTSQPVA IKKIMKPFST 60
61 AVLAKRTYRE LKLLKHLRHE NLICLQDIFL SPLEDIYFVT ELQGTDLHRL LQTRPLEKQF 120
121 VQYFLYQILR GLKYVHSAGV IHRDLKPSNI LINENCDLKI CDFGLARIQD PQMTGYVSTR 180
181 YYRAPEIMLT WQKYDVEVDI WSAGCIFAEM IEGKPLFPGK DHVHQFSIIT DLLGSPPKDV 240
241 INTICSENTL KFVTSLPHRD PIPFSERFKT VEPDAVDLLE KMLVFDPKKR ITAADALAHP 300
301 YSAPYHDPTD EPVADAKFDW HFNDADLPVD TWRVMMYSEI LDFHKIGGSD GQIDISATFD 360
361 DQVAAATAAA AQAQAQAQAQ VQLNMAAHSH NGAGTTGNDH SDIAGGNKVS DHVAANDTIT 420
421 DYGNQAIQYA NEFQQ |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
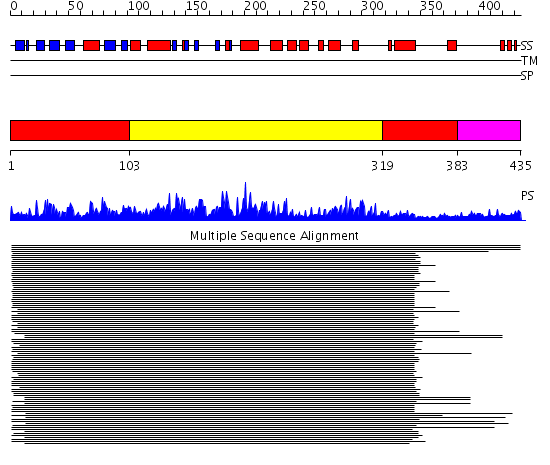
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..102] [319..382] |
1185.0 | MAP kinase p38 | |
| 2 | View Details | [103..318] | 1185.0 | MAP kinase p38 | |
| 3 | View Details | [383..435] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.98 |
Source: Reynolds et al. (2008)