













| General Information: |
|
| Name(s) found: |
Fps85D-PA /
FBpp0081530
[FlyBase]
|
| Description(s) found:
Found 22 descriptions. SHOW ALL |
|
| Organism: | Drosophila melanogaster |
| Length: | 1325 amino acids |
Gene Ontology: |
|
| Cellular Component: |
adherens junction
[IDA]
cytoplasm [NAS] membrane [NAS] plasma membrane [IDA] |
| Biological Process: |
dorsal closure
[IGI]
photoreceptor cell morphogenesis [IMP] dorsal closure, elongation of leading edge cells [IMP] axon guidance [IMP] dorsal closure, amnioserosa morphology change [IMP] protein amino acid phosphorylation [IEA][NAS] actin filament bundle assembly [IMP] |
| Molecular Function: |
ATP binding
[IEA]
protein tyrosine kinase activity [ISS][NAS] protein binding [IEA] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MGFSSALQSR AAHEALIVRQ DAELRLMETM KRSIQMKAKC DKEYAISLTA VAQQGLKIDR 60
61 ADEMQGSLIS KSWRSYMDEL DHQAKQFKFN AEQLEVVCDK LTHLSQDKRK ARKAYQEEHA 120
121 KIAARLNHLT DEVVRKKSEY QKHLEGYKAL RTRFEENYIK APSRSGRKLD DVRDKYQKAC 180
181 RKLHLTHNEY VLSITEAIEV EKDFRNVLLP GLLEHQQSVQ ESFILLWRNI LQEAAQYGDL 240
241 TADKYKEIQK RIDTVIGSIN PTEEYGEFTE KYKTSPTTPL LFQFDETLIQ DIPGKLQSST 300
301 LTVDNLTVDW LRNRLQELEG AVRDCQEKQM KMIEHVNGGS PVANGSIISN GSNTSNGIQS 360
361 NKDSLCRQSK DLNALRCQEK QKQKLVDMIK CALNEVGCEE LPSGCDDDLT LEQNFIENGY 420
421 NNEQQRSNST SSPGLGIMNE LMRRGGVLTL LRGRGRHFKR KSTPQPATPM TRSRQGRFNK 480
481 LQPRSQSLGS LSVIRDGNGP SPARYEPITN HRLRQAASVH YLGEEIATSS TNPPDLTRLR 540
541 RTQCSMLCLG EDEEPVVLAS PAPLTQLTAA VLTNTNNNHI YADLELDKKK DTSPSPECKG 600
601 EQIQPKKEQI RIEINQTAPQ NSIDAHLDRI DELNRVLDDR LKRTLQPSDD VNAIESAEEN 660
661 HIQTRKLAKD PDSQTKRSSS SSSECRSSKD TSHSKKRSLS FSQKSISNIF SNLKEFSKSP 720
721 LVRMGKNHIL NEEQDAKRTQ PSQHHHSSGS DCPTNSSSSS SNNNNNNKNT SSNSNHSASQ 780
781 STIITSTITT TITTTTTTTP SKENSRLKFK VPKIQKKSKA IRNTFRSKLL NFQLKRSKPC 840
841 KQCTKRRRIH PSKSVFDFAK EFEVEQPAGS AADEQFCNCP PAGQKPVKPS VQISGHKDHP 900
901 FESSSGELDE NSDRDIDNDE EEEDSASDDV LSMKDHCYCV PSLAASISLS TNRPLYEEEW 960
961 FHGVLPREEV VRLLNNDGDF LVRETIRNEE SQIVLSVCWN GHKHFIVQTT GEGNFRFEGP1020
1021 PFASIQELIM HQYHSELPVT VKSGAILRRP VCRERWELSN DDVVLLERIG RGNFGDVYKA1080
1081 KLKSTKLDVA VKTCRMTLPD EQKRKFLQEG RILKQYDHPN IVKLIGICVQ KQPIMIVMEL1140
1141 VLGGSLLTYL RKNSNGLTTR QQMGMCRDAA AGMRYLESKN CIHRDLAARN CLVDLEHSVK1200
1201 ISDFGMSREE EEYIVSDGMK QIPVKWTAPE ALNFGKYTSL CDVWSYGILM WEIFSKGDTP1260
1261 YSGMTNSRAR ERIDTGYRMP TPKSTPEEMY RLMLQCWAAD AESRPHFDEI YNVVDALILR1320
1321 LDNSH |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
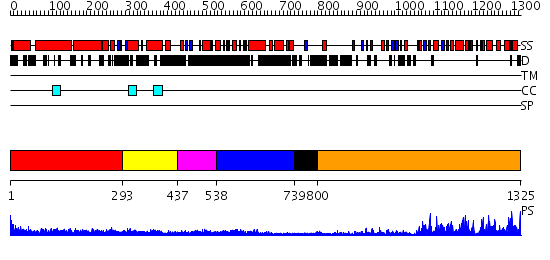
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..292] | 26.69897 | No description for 2eflA was found. | |
| 2 | View Details | [293..436] | 5.0 | Tropomyosin | |
| 3 | View Details | [437..537] | 2.49 | Solution structure of the human FES SH2 domain | |
| 4 | View Details | [538..738] | 1.321994 | View MSA. No confident structure predictions are available. | |
| 5 | View Details | [739..799] | N/A | No confident structure predictions are available. | |
| 6 | View Details | [800..1325] | 121.0 | No description for 2h8hA was found. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.98 |
Source: Reynolds et al. (2008)