













| General Information: |
|
| Name(s) found: |
mde3 /
SPBC8D2.19
[Sanger Pombe]
|
| Description(s) found:
Found 19 descriptions. SHOW ALL |
|
| Organism: | Schizosaccharomyces pombe |
| Length: | 559 amino acids |
Gene Ontology: |
|
| Cellular Component: |
cellular_component
[ND]
|
| Biological Process: |
ascospore formation
[IGI signal transduction [RCA protein amino acid phosphorylation [ISS |
| Molecular Function: |
ATP binding
[ISS protein serine/threonine kinase activity [ISS |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSNESIYSVL DLTQVIFEDR YLVKQKLGDG SFGTVYLAQR KEKNGLYETV AVKKLKNSSK 60
61 PKPKHELLKL RESLALRKIS KHPCLIDLLE TFMDPYRNIF LVMEFMDCNL FQLFKRRQGR 120
121 LFTKETAFNI LLQIISGIEH IHKHGFMHRD IKPENILVKR ISPKPISSRY SIKLGDFGLA 180
181 RPSVSSDPLT EYVSTRWYRA PELLLRSGSY NHSVDLYAFG CIVFEIYSLK PLFPGRNETD 240
241 QLNRVCEILG NPGIDELDTL HYWSQAKELA KRLGFMLPPT KPYPIQKLLP QNCPEGHAKM 300
301 IPCLLAWNPD VRPTAKYCKE VFFPLPPSAS KSNSVPQKIS NPKVEQNLGF PISREDKKST 360
361 RRVGWLKKNL SEFVSSVKSV FPDSHGSQPH VKTEKPINAK ESTGHLANPI ASSNVPAISL 420
421 KPGELHESVF FSENEQIDYL LTSIDYLPSY KPPSNGSNIA INAFNETVGD RIPSSKDILI 480
481 TEKIPFKKEN EIRDSIVPSC SQPDESNKEG VASCLLLQKS GMEMTSVLEY STPNPAEVQN 540
541 ICNDHAKFET SKSLHLSSP |
The following runs contain data for this protein:
| BAIT | COMMENTS | PUBLICATION | |
| View Run | No Comments | Liu X, et al. (2005) |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
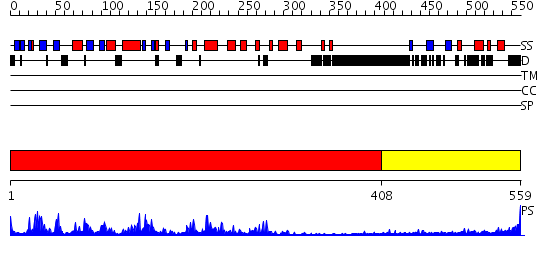
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..407] | 66.0 | MAP kinase p38 | |
| 2 | View Details | [408..559] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.97 |
Source: Reynolds et al. (2008)