













| General Information: |
|
| Name(s) found: |
slik-PD /
FBpp0072273
[FlyBase]
slik-PB / FBpp0072271 [FlyBase] |
| Description(s) found:
Found 37 descriptions. SHOW ALL |
|
| Organism: | Drosophila melanogaster |
| Length: | 1703 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: |
cell proliferation
[IMP]
regulation of localization [IMP] cytokinesis [IMP] positive regulation of peptidyl-threonine phosphorylation [IMP] protein amino acid phosphorylation [IDA][NAS] |
| Molecular Function: |
polo kinase kinase activity
[NAS]
ATP binding [IEA] receptor signaling protein serine/threonine kinase activity [ISS][NAS] protein serine/threonine kinase activity [IDA] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSFITNLKKV FHLGGGEAKK KRLYNNIKMD TDPAEFWEMV GELGDGAFGK VYKAQHKEQK 60
61 RFAAAKMCQL EDEENLSDHM VEIDILSEIK HPNIVELYEA FSIDDKLWML IEYCDGGALD 120
121 SIMVELEKPL TEPQIAYVCK HMTEGLTFLH RNKVIHRDLK AGNVLLTMEG GVKLADFGVS 180
181 AKNKHTMQKH DTFIGTPYWM APELVLCETF RDNPYDHKVD IWSLGITLIE LAQMEPPNSE 240
241 MSPMRVLLKI QKSEPPKLEQ PSRWSKEFND FLKKSLVKDP QVRPTTDVLM QHAFINRNLD 300
301 AKPIKDLLLE YKAEVVEEVV DDETEEPRNS ALQLDLDDDS ASLQSQDIDK LPGTPTSILR 360
361 DAKEQSQPSS SLPIAAAATA AAAATTTTKA TTPDRPNHTK DDNAEAAAQQ PPHTKVPAPA 420
421 PPSSQQTPPP QVQQPPTPPA QPTAAVLQKK PEDVAAVAET SEKTESDKKH FVKKGKAPPP 480
481 PSPLGLANAK PAASDSQTSP KKLATPEPTS PVTTAIEVAI GQEAMEPKPQ PPSPTASSIV 540
541 SVQSVASSSS SGSVSNAVLS SSTSLITINS DPPTPHHHQP LPPQPQHLIL PNSLESVSQI 600
601 TVVTSTHPPV IIDNSVMPPQ NEVIIVSNDM NKSTHLHESS TDDDFPSLDD SLGDATTPPH 660
661 KQSSMILAVN EPAGVVPAPP SQPQTSSTVH ARKLDESEVL IVSPSYADDD SAYNTASGSH 720
721 SHDHSDHLMD TSHVSVVTVG DEGVKVKDSS NELVKRQPNG VGIVPEDVNI IVNRFKQEKR 780
781 SPDSSLSENG SVRGRRGIEV LVGGSGGSDV DSIGTNTSQE SRHETDHNNK QQYPAAALMP 840
841 PPPPSLTNNH NHETIDEEEE VVIRPKARVP AVVKSANAQG LTKEEIELRN LRKKTRKRTR 900
901 KFEIDGVQMT TTTSRVIYGD DENGRIYDDH DFRKQELREL KLLQKQEKKQ QTELHLKEQQ 960
961 AKEQQDRRFE QERSSLEKTY EADMDMLARQ HKQLVEKTEQ TQENELRSSS KRIRSEQEQE1020
1021 LKIFRENLKQ EIRLLKQEVD LFPKDKRKDE FKQRRSAMEL DHEEKERAFL DSLKERHELL1080
1081 LRRLSEKHRD HLATINRNFL QQKQNAMRTR EALLWELEEK QLHERHQLSK RHVKELCFMQ1140
1141 RHQMIIRHEK ELDQVKRMLQ RKEEDMVKKQ TMEKRALPKR IRAERKARDL MFRESLRIST1200
1201 NLDPEIERDR LKKFQEQEKK RYMQEERRFE VKHQKQLEEL RATRESAIKE LEQLQNEKRR1260
1261 ALVEHEHSKL SEIDERLKGE LREWREQLVP RKQELNRQIK LAIDQHEKRF GLVTNREEFE1320
1321 DQEVKLPAHL RNIYNERSSR ILPRNTFIDP QNMPRSRSSL LMMGGGGNRL SNFRGSAPDL1380
1381 SRSVPNTPIM HKQQRSQMIG DQVLEEDEEQ MLSEQMKRAS VATLPAQNQL RLITQLPANE1440
1441 LVKVVTSTPP LNSAEDVNSK PSMSTFRGST PPKTPDDLGL VTNRFSRLDT KAAAEASGVE1500
1501 LRIHEGPVLT AHTQRLLHTT SFAIKRPSLA SRSSGRSSLS SAQSMYNINE LSTRFTSDAD1560
1561 LIFDSGRKAA ALLDEVQPQL RSSVKPGHSR ANGSVVSADS AASSEDIYYD LYAAQYKLPR1620
1621 QLQQRLQQEV SPLIYPQCAS QPFDTTQGPE TGARRLEETF AQQLDEMETL YGGALIVSMP1680
1681 SDTLQRDHFT GSTRSSLSSY SEG |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
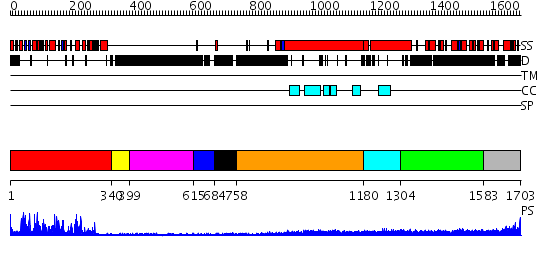
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..339] | 69.69897 | No description for 2j51A was found. | |
| 2 | View Details | [340..398] | N/A | No confident structure predictions are available. | |
| 3 | View Details | [399..614] | N/A | No confident structure predictions are available. | |
| 4 | View Details | [615..683] | N/A | No confident structure predictions are available. | |
| 5 | View Details | [684..757] | N/A | No confident structure predictions are available. | |
| 6 | View Details | [758..1179] | 19.522879 | Heavy meromyosin subfragment | |
| 7 | View Details | [1180..1303] | 1.49 | crystal structure of the cytotoxic bacterial protein colicin B at 2.5 A resolution | |
| 8 | View Details | [1304..1582] | 8.0 | Tropomyosin | |
| 9 | View Details | [1583..1703] | 3.221849 | Crystal Structure Analysis of ClpB |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 8 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 9 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)