













| General Information: |
|
| Name(s) found: |
pck2 /
SPBC12D12.04c
[Sanger Pombe]
|
| Description(s) found:
SHOW ONLY BEST |
|
| Organism: | Schizosaccharomyces pombe |
| Length: | 1016 amino acids |
Gene Ontology: |
|
| Cellular Component: |
cell septum
[IDA cell division site [IDA cell tip [IDA |
| Biological Process: |
positive regulation of calcium-mediated signaling
[IMP regulation of cell shape [IEA] regulation of establishment or maintenance of cell polarity [IMP regulation of alpha-glucan biosynthetic process [IMP protein amino acid phosphorylation [IC regulation of 1,3-beta-glucan biosynthetic process [IMP regulation of fungal-type cell wall biogenesis [IMP |
| Molecular Function: |
ATP binding
[IEA]
zinc ion binding [IEA] protein binding [IPI protein kinase C activity [TAS |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MDMIDEAITE VVRKIERERS VIHGALSMKR LTQNQTVHQQ LHSNIEESKK SIIYLEERLE 60
61 KLKLRKNGVR KSNSEKPSVG IEKNPSFSTT KSAKSFSSTS SNIDSNLDLL NYDTPLTISK 120
121 ISFLLQQLEF KLSVEEQYRK GIEKMAKLYE REHDRRSIAE AEKKYVESAQ KITLLKQALK 180
181 RYHDLHIEID EEDVPSTESR GNLNARRPQS GLLKITVGSL RNVTHSAGIS KQTEMIVAIR 240
241 AEDLERARTR PSRTDRFNET FEIDLEKTNE VEIVVYEKKN EKLLLPVGLL WIRLSDLVEK 300
301 QRRKKVEQEV SDKGWVSADK MINQRLSIFL PSALNNISKP ESTDRPNTAS GNQSVSAWFS 360
361 LEPMGQINLT MNFTKHNTRK RPMDAGLGRQ GAIRQRKESV HEVYGHKFLQ HQFYQIMRCA 420
421 LCGEFLKNAA GMQCIDCHYT CHKKCYPKVV TKCISKSSDS ASSEYEKINH RIPHHFESHT 480
481 NIGANWCCHC GYILPLGRKT ARKCTECGIT AHAQCVHLVP DFCGMSMEMA NRVISEIRTT 540
541 KIYKAQQHKQ KSSHHKHHHH KKSKSSSSKH KENDKASVSI TTTTTPSITP ADPVPTSPKP 600
601 LAIEPVKRKP VHAGNLEVTS VSDNKLGATV QVVEQKVDDK ADALTKPPSL DAVKEPIPVP 660
661 SVETSVVAQD LTHKAKRIGL EDFTFLSVLG KGNFGKVMLA ELKSEKQLYA IKVLKKEFIL 720
721 ENDEVESTKS EKRVFLVANR ERHPFLVNLH SCFQTETRIY FVMDFVSGGD LMLHIQQEQF 780
781 SRRRAQFYAA EVCLALKYFH DNGIIYRDLK LDNILLSPDG HVKVADYGLC KEDMWHDNTT 840
841 ATFCGTPEFM APEILLEQQY TRSVDWWAFG VLIYQMLLGQ SPFRGEDEEE IFDAILSDEP 900
901 LYPIHMPRDS VSILQQLLTR DPKKRLGSGP NDAEDVMTHP FFSNINWDDI YHKRTQPPYI 960
961 PSLNSPTDTK YFDEEFTREL PVLTPVNSIL TKEMQQHFEG FSYSCEDDKP STTDNA |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
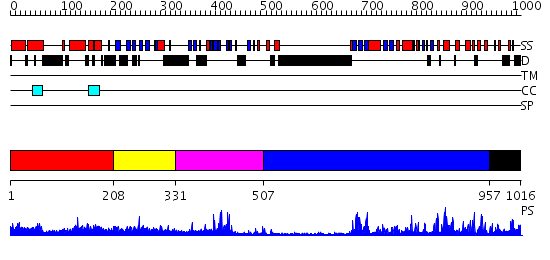
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..207] | 5.30103 | Heavy meromyosin subfragment | |
| 2 | View Details | [208..330] | 18.69897 | Domain from protein kinase C epsilon | |
| 3 | View Details | [331..506] | 14.522879 | Crystal Structure of the Human Beta2-Chimaerin | |
| 4 | View Details | [507..956] | 62.39794 | Abl tyrosine kinase, SH3 domain; Abl tyrosine kinase; Abelsone tyrosine kinase (abl) | |
| 5 | View Details | [957..1016] | 95.221849 | Pkb kinase |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.98 |
Source: Reynolds et al. (2008)