













| General Information: |
|
| Name(s) found: |
Khc-73-PA /
FBpp0086490
[FlyBase]
|
| Description(s) found:
Found 28 descriptions. SHOW ALL |
|
| Organism: | Drosophila melanogaster |
| Length: | 1921 amino acids |
Gene Ontology: |
|
| Cellular Component: |
kinesin complex
[ISS]
astral microtubule [IDA] |
| Biological Process: |
establishment of spindle orientation
[IMP]
microtubule-based movement [ISS] |
| Molecular Function: |
ATP binding
[IEA]
microtubule motor activity [ISS] microtubule binding [ISS] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MASDKIKVAV RVRPFNRREI ELDTKCIVEM EKQQTILQNP PPLEKIERKQ PKTFAFDHCF 60
61 YSLNPEDENF ASQETVFDCV GRGILDNAFQ GYNACIFAYG QTGSGKSYTM MGTQESKGII 120
121 PRLCDQLFSA IANKSTPELM YKVEVSYMEI YNEKVHDLLD PKPNKQSLKV REHNVMGPYV 180
181 DGLSQLAVTS YQDIDNLMTE GNKSRTVAAT NMNAESSRSH AVFSVVLTQI LTDQATGVSG 240
241 EKVSRMSLVD LAGSERAVKT GAVGDRLKEG SNINKSLTTL GLVISKLADQ SNGKKSGNDK 300
301 FVPYRDSVLT WLLKDNLGGN SRTVMVATIS PSADNYEETL STLRYADRAK RIVNHAVVNE 360
361 DPNARIIREL RHEVETLRSM LKHATGSPVG DVQDKLAESE NLMKQISQTW EEKLVKTERI 420
421 QNERQQALEK MGISVQASGI KVEKNKYYLV NLNADPSLNE LLVYYLKDRT LIGGRTISGQ 480
481 QPDIQLSGLG IQPEHCVITI EDSGLYMEPV QGARCFVNGS AAVEKTPLQN GDRILWGNHH 540
541 FFRVNSPKSN NTSMCASEPQ TPAQLIDYNF ARDEIMQNEL SNDPIQTAIA RLERQHEEDK 600
601 QVALEKQRQE YERQFQQLRN ILSPSTPYAP YAPYDPLRMG KITPNTPTSQ MRVEKWAQER 660
661 DEMFRRSLGQ LKTDIMRANS LVQEANFLAE EMEKKTKFSV TLQIPPANLS PNRRRGAFVS 720
721 EPAILVKRTN SGSQIWTMEK LENKLIDMRE MYQEHKERVL NGLPLIEPFS EDEFDDKDED 780
781 NAKPQDPFYE SQENHNLIGV ANIFLEVLFH DVKLDYHTPI ISQQGEVAGR LQVEIERIAG 840
841 QMPQDRMCES VSESSGDSRD EYDDPVDPTS NQITCRVTIK CASGLPLSLS NFVFCQYTFW 900
901 GHQEMVVPVI NAESTAHDQN MVFKFEHTQD FTVTINEEFL EHCIEGALSI EVWGHRSAGF 960
961 SKTKGWEVEQ QQAKARSLVD RWAELSRKIE LWVEIHELND NGEYSPVEVT NRNEVLTGGI1020
1021 YQLRQGQQRR VNVRVKPVQN SGTLPIICQS IVNVAIGSVT VRSRLQRPLD SYQEEDLTVL1080
1081 REKWSEALGR RRQYLDQQIQ MLIKKEEKNE QERERELSLV HQWVSLTEER NAVLVPAPGS1140
1141 GIPGAPASWE PPSGMEPHVP VLFLNLNGDD LSAQNTNDEL SVAGINSILS KEHGHKFYTL1200
1201 QILQHLDKDV CCVASWDSSM HDSQALNRVT EANERVYLIL RTTVRLSHPA PMDLVLRKRL1260
1261 SINIKKGQTL TDRLKKFRLV RGENAIWQSG VTYEVVSNIP KASEELEDRE SLAQLAASGD1320
1321 DCSASDGETY IEKYTRGVSA VESILTLDRL RQNVAVKELE TAHGQPLSMR KTVSVPNFSQ1380
1381 QLINKLTQIM RFDASMESLL NVGRSESFAD LNNSALGNKF TPAGHSPAGA GGVIRSRHSF1440
1441 GGKGSSDDSP GKAFGIASPA TSKLLGMRMT TLHEEPLGGH RSLDEEPEDS YSDSEYAAEY1500
1501 EQERQQNKSM ATRSRLTASK TMDSFMDVSS HSNQSYLSYT SSANANMKHL TGLATLSMSS1560
1561 STSSGYGSQA VSCNNLSNED IASMRSMSID ETPDFDRVNS NSPPNRQARV NPFLKDMPKA1620
1621 KIQEQPEQQA KKLQEAFTHP LEQLESSQNA QSDDDECAQL PKNNNNNLDL VNEPKPLSSQ1680
1681 TDLEESMSQP KSKTEFATDN QNGNRSSDEL SHSSEDLLEG DGIVREELPA GKVVRRKKSN1740
1741 TQPPSNGNSI NNNNNGTTQA PRINHRASVA KMEGLAAYLD SSIMTSSTEV DEESKDVELV1800
1801 LPEWIVVGES VLIRPYNTSG VIRFVGTTEF QPGAWIGVEL DTPTGKNDGS VKGVQYFQCK1860
1861 PKHGMFVRSD KLMLDKRGKA MRAYKAAEKS NSISKEMSTS MTGSMTRSKS RGDSLNLSAR1920
1921 K |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
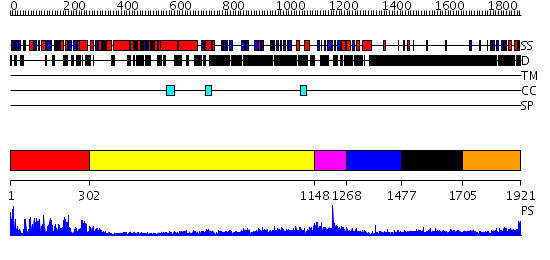
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..301] | 118.0 | Kinesin | |
| 2 | View Details | [302..1147] | 14.69897 | Heavy meromyosin subfragment | |
| 3 | View Details | [1148..1267] | 1.149994 | View MSA. No confident structure predictions are available. | |
| 4 | View Details | [1268..1476] | N/A | No confident structure predictions are available. | |
| 5 | View Details | [1477..1704] | 3.331996 | View MSA. No confident structure predictions are available. | |
| 6 | View Details | [1705..1921] | 11.09691 | Solution structure of the 2nd CAP-Gly domain in human CLIP-170/restin |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)