













| General Information: |
|
| Name(s) found: |
PRR2_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 699 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSLSRILRYN QRNNKTTASL TAEHAYSDNW AYSVSLGDPT SVGVNMAAKT GEALNKSYDS 60
61 VFSSLPVADS VPRTDFTASS RDDENTDVQK LTTSWMEKID TKMPENISKI DSNIISSPMV 120
121 SKVEARFIVP KGRLRKNSTD FTSSFSNSLS LPKSYGKLIF FTSKKNSSST KKNLANDISD 180
181 NKHNNNSSNT IGHNIPVTTA TATCDEIACT STEHEYNVYE EERMFTTRVY SLEDSVSSLS 240
241 TNPLDDTYSE AVQVNTRHIE DTESTAHIRK HSYTTSLSSI KRLFKITSFS NNNSNSCDHQ 300
301 ESTVADDCAI SSSLKETTSS PVSTGSFSLM IENEDSDRDQ IIQALYSNIE ASTDLVSRKY 360
361 RDLDVVLGEG SGGKVKLVQR VLDNKVFALK EYRSKKKRES ERKYIKNIIS EYCIASTLKN 420
421 PNICETLEIL YEKGKIFQIL EYCEYDLFSL VMSEKMHYEE ICCLFKQLIN GVKYLHDIGL 480
481 SHRDLKLDNC VVTRRGILKL IDFGASSVFH YPLSSQMIEA NGIVGSDPYL SPEVFYFNEY 540
541 DPRALDVWSV GIIFFCMITR RFPWKYPKVK DVQFKAFCSG RGVSSFKDLV TRPATDDSNN 600
601 YDNDGYEEGV IDMGPNFILH RLPEETHKIM RRILEVSPFR RITINGILQD GWIKEIETCQ 660
661 VVGAASPNEA SLRIINKGNH IHTNIDQRYA HIGGLHQRT |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
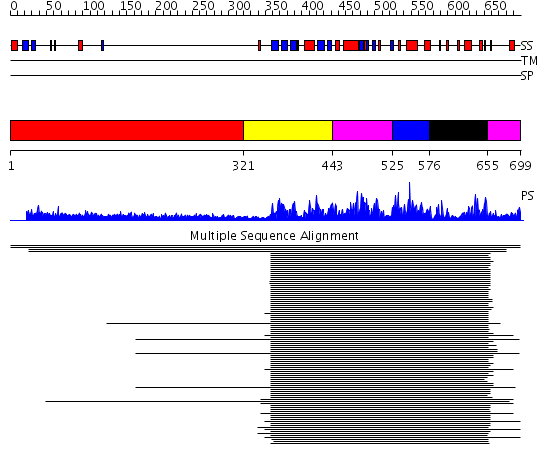
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..320] | N/A | No confident structure predictions are available. | |
| 2 | View Details | [321..442] | 319.228787 | Cyclin-dependent PK, CDK2 | |
| 3 | View Details | [443..524] [655..699] |
319.228787 | Cyclin-dependent PK, CDK2 | |
| 4 | View Details | [525..575] | 319.228787 | Cyclin-dependent PK, CDK2 | |
| 5 | View Details | [576..654] | 319.228787 | Cyclin-dependent PK, CDK2 |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.77 |
Source: Reynolds et al. (2008)