













| General Information: |
|
| Name(s) found: |
CDC7_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 507 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MTSKTKNIDD IPPEIKEEMI QLYHDLPGIE NEYKLIDKIG EGTFSSVYKA KDITGKITKK 60
61 FASHFWNYGS NYVALKKIYV TSSPQRIYNE LNLLYIMTGS SRVAPLCDAK RVRDQVIAVL 120
121 PYYPHEEFRT FYRDLPIKGI KKYIWELLRA LKFVHSKGII HRDIKPTNFL FNLELGRGVL 180
181 VDFGLAEAQM DYKSMISSQN DYDNYANTNH DGGYSMRNHE QFCPCIMRNQ YSPNSHNQTP 240
241 PMVTIQNGKV VHLNNVNGVD LTKGYPKNET RRIKRANRAG TRGFRAPEVL MKCGAQSTKI 300
301 DIWSVGVILL SLLGRRFPMF QSLDDADSLL ELCTIFGWKE LRKCAALHGL GFEASGLIWD 360
361 KPNGYSNGLK EFVYDLLNKE CTIGTFPEYS VAFETFGFLQ QELHDRMSIE PQLPDPKTNM 420
421 DAVDAYELKK YQEEIWSDHY WCFQVLEQCF EMDPQKRSSA EDLLKTPFFN ELNENTYLLD 480
481 GESTDEDDVV SSSEADLLDK DVLLISE |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
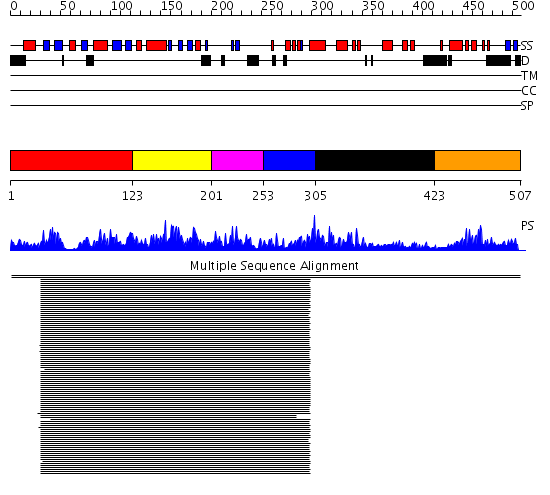
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..122] | 243.0 | Cyclin-dependent PK, CDK2 | |
| 2 | View Details | [123..200] | 243.0 | Cyclin-dependent PK, CDK2 | |
| 3 | View Details | [201..252] | 243.0 | Cyclin-dependent PK, CDK2 | |
| 4 | View Details | [253..304] | 243.0 | Cyclin-dependent PK, CDK2 | |
| 5 | View Details | [305..422] | 1.322978 | View MSA. No confident structure predictions are available. | |
| 6 | View Details | [423..507] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.97 |
Source: Reynolds et al. (2008)