













| General Information: |
|
| Name(s) found: |
ENSCJAT00000010341
[marm_p_042810_contam.fasta]
|
| Description(s) found: |
|
| Organism: | Callithrix jacchus |
| Length: | 740 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MPLAQLADPW QKMAVESPSD SAENGQQIMD EPMGEEEINP QTEEVSIKEI AITHHVKEGH 60
61 EKADPSQFEL LKVLGQGSFG KVFLVKKISG SDARQLYAMK VLKKATLKVR DRVRTKMERD 120
121 ILVEVNHPFI VKLHYAFQTE GKLYLILDFL RGGDLFTRLS KEVMFTEEDV KFYLAELALA 180
181 LDHLHSLGII YRDLKPENIL LDEEGHIKLT DFGLSKESID HEKKAYSFCG TVEYMAPEVV 240
241 NRRGHTQSAD WWSFGVLMFE MLTGTLPFQG KDRKETMTMI LKAKLGMPQF LSPEAQSLLR 300
301 MLFKRNPANR LGAGPDGVEE IKRHSFFSTI DWNKLYRREI HPPFKPATGR PEDTFYFDPE 360
361 FTAKTPKDSP GIPPSANAHQ LFRGFSFVAI TSDDESQAMQ TVGVHSIVQQ LHRNSIQFTD 420
421 GYEVKEDIGV GSYSVCKRCI HKATNMEFAV KIIDKSKRDP TEEIEILLRY GQHPNIITLK 480
481 DVYDDGKYVY VVTELMKGGE LLDKILRQKF FSEREASAVL FTITKTVEYL HAQGVVHRDL 540
541 KPSNILYVDE SGNPESIRIC DFGFAKQLRA ENGLLMTPCY TANFVAPEVL KRQGYDAACD 600
601 IWSLGVLLYT MLTGYTPFAN GPDDTPEEIL ARIGSGKFSL SGGYWNSVSD TAKDLVSKML 660
661 HVDPHQRLTA ALVLRHPWIV HWDQLPQYQL NRQDAPHLVK GAMAATYSAL NRNQSPVLEP 720
721 VGRSTLAQRR GIKKITSTAL |
SHOWING SINGLE HITS. [ Hide Single Hits ]
New Feature: Upload Your Own Microscopy Data
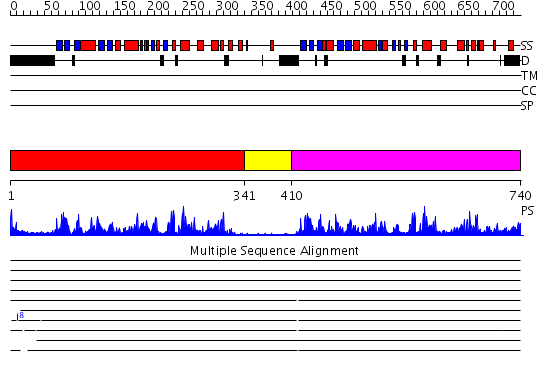
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..340] | 78.045757 | The structure of the N-terminal kinase domain of MSK1 reveals a novel autoinhibitory conformation for a dual kinase protein | |
| 2 | View Details | [341..409] | 2.03 | Crystal Structure of ROCK 1 bound to fasudil | |
| 3 | View Details | [410..740] | 91.522879 | crystal structure of an inactive Akt2 kinase domain |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.96 |
Source: Reynolds et al. (2008)