













| General Information: |
|
| Name(s) found: |
M3K3_ARATH
[Swiss-Prot]
|
| Description(s) found:
SHOW ONLY BEST |
|
| Organism: | Arabidopsis thaliana |
| Length: | 651 amino acids |
Gene Ontology: |
|
| Cellular Component: |
apoplast
[IDA]
|
| Biological Process: |
cortical microtubule organization
[IMP]
|
| Molecular Function: |
ATP binding
[IEA]
protein kinase activity [IEA] protein serine/threonine kinase activity [IEA] kinase activity [ISS] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MQDILGSVRR SLVFRSSLAG DDGTSGGGLS GFVGKINSSI RSSRIGLFSK PPPGLPAPRK 60
61 EEAPSIRWRK GELIGCGAFG RVYMGMNLDS GELLAIKQVL IAPSSASKEK TQGHIRELEE 120
121 EVQLLKNLSH PNIVRYLGTV RESDSLNILM EFVPGGSISS LLEKFGSFPE PVIIMYTKQL 180
181 LLGLEYLHNN GIMHRDIKGA NILVDNKGCI RLADFGASKK VVELATVNGA KSMKGTPYWM 240
241 APEVILQTGH SFSADIWSVG CTVIEMATGK PPWSEQYQQF AAVLHIGRTK AHPPIPEDLS 300
301 PEAKDFLMKC LHKEPSLRLS ATELLQHPFV TGKRQEPYPA YRNSLTECGN PITTQGMNVR 360
361 SSINSLIRRS TCSGLKDVCE LGSLRSSIIY PQKSNNSGFG WRDGDSDDLC QTDMDDLCNI 420
421 ESVRNNVLSQ STDLNKSFNP MCDSTDNWSC KFDESPKVMK SKSNLLSYQA SQLQTGVPCD 480
481 EETSLTFAGG SSVAEDDYKG TELKIKSFLD EKAQDLKRLQ TPLLEEFHNA MNPGIPQGAL 540
541 GDTNIYNLPN LPSISKTPKR LPSRRLSAIS DAMPSPLKSS KRTLNTSRVM QSGTEPTQVN 600
601 ESTKKGVNNS RCFSEIRRKW EEELYEELER HRENLRHAGA GGKTPLSGHK G |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
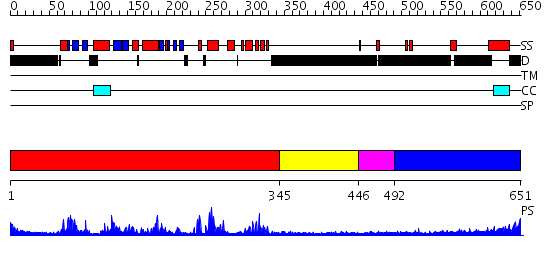
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..344] | 61.39794 | No description for 2j51A was found. | |
| 2 | View Details | [345..445] | N/A | No confident structure predictions are available. | |
| 3 | View Details | [446..491] | N/A | No confident structure predictions are available. | |
| 4 | View Details | [492..651] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.89 |
Source: Reynolds et al. (2008)