













| General Information: |
|
| Name(s) found: |
RH25_ARATH
[Swiss-Prot]
|
| Description(s) found:
Found 15 descriptions. SHOW ALL |
|
| Organism: | Arabidopsis thaliana |
| Length: | 563 amino acids |
Gene Ontology: |
|
| Cellular Component: |
cellular_component
[ND]
|
| Biological Process: |
response to cold
[IEP]
response to salt stress [IMP] response to water deprivation [IMP] |
| Molecular Function: |
RNA binding
[IDA]
ATP-dependent helicase activity [ISS] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MNSDGPKSGK KRREIRAKLV KKLTSDEDGS GKLVKDNNKS LKRGREGKSD VDEPLIKKPA 60
61 STTPLVTQIA KTSDSYLSKT RFDQFPLSPL TLKGIEDAGF KTMTVVQEAT LPLILQGKDI 120
121 LAKAKTGTGK TVAFLLPSIE AVIKAPPASR DNRHPPIIVL VVCPTRELAC QAAAEANILL 180
181 KYHPSIGVQV VIGGTKLPTE QRRLQKSPCQ ILVATPGRLK DHIDNTSGFA TRLMGVKVLV 240
241 LDEADHLLDM GFRREIERII AAVPKQRQTF LFSATVSDEV RQICHVALKR DHEFVNCVQE 300
301 GAGETHQKVS QMYMIASLDR HFSLLYGLLK KHITDNVGYK VIIFCTTAMV TRLVADLLGK 360
361 LSLNVREIHS RKPQSYRTRV SDEFRKSKSI ILVTSDVSAR GVDYPDVSLV VQMGLPSDRE 420
421 QYIHRLGRTG RKGKEGEGVL LLAPWEEYFL SSVKDLPITK SSLPPIDHEA VKKVQKGLIQ 480
481 VEMTNKEAAY QAWLGYYKSQ KKIARDTTRL VELANEFSRS MGLSIPPAIP VNILGKMGLK 540
541 NVPGIRVAPG FDKKPAKRNY RSR |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
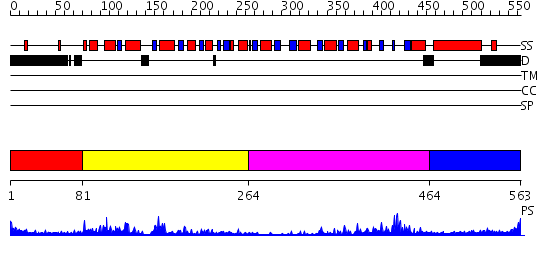
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..80] | 73.69897 | No description for 2db3A was found. | |
| 2 | View Details | [81..263] | 5.69897 | No description for 2fz4A was found. | |
| 3 | View Details | [264..463] | 7.3 | No description for 2p6nA was found. | |
| 4 | View Details | [464..563] | 26.39794 | Nucleotide excision repair enzyme UvrB |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.97 |
Source: Reynolds et al. (2008)