













| General Information: |
|
| Name(s) found: |
AGC15_ARATH
[Swiss-Prot]
|
| Description(s) found:
SHOW ONLY BEST |
|
| Organism: | Arabidopsis thaliana |
| Length: | 577 amino acids |
Gene Ontology: |
|
| Cellular Component: |
chloroplast
[IEA]
|
| Biological Process: |
unidimensional cell growth
[IGI]
pollen tube growth [IGI] |
| Molecular Function: |
kinase activity
[ISS]
|
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MDLASKKNTA NVGSKEIDPI KPKSPRSSLS PFSLKLGDNV PRNPHFDPKK MDPLVKHQPP 60
61 KSLEPPPSTR GTNSEGDLKH NTYSSDGDSL AMRKNAPKNL HYDPKKIVPL TTSETYSPSA 120
121 RNHHHHRTKS PDKKRAPRHN GDYAYGDNLV GPSAQPFKPH TGGDVRWDAI NSIASKGPQI 180
181 GLDNFRLLKR LGYGDIGSVY LADLRGTNAV FAMKVMDKAS LASRNKLLRA QTEREILSLL 240
241 DHPFLPTLYS YFETDKFYCL VMEFCSGGNL HSLRQKQPSR RFTEEAARFY ASEVLLALEY 300
301 LHMLGVVYRD LKPENILVRD EGHIMLSDFD LSLRCTFNPT LVKSSSVCSG GGAILNEEFA 360
361 VNGCMHPSAF LPRLLPSKKT RKAKSDSGLG GLSMPELMAE PTDVRSMSFV GTHEYLAPEI 420
421 IRGEGHGSAV DWWTFGIFLY ELLHGTTPFK GQGNRATLHN VVGQPLKFPD TPHVSSAARD 480
481 LIRGLLVKDP HRRIAYTRGA TEIKQHPFFE GVNWALVRSA APPHIPDPVD LGPYAAARGK 540
541 TKSHGGGDHC NSMKPEPLVA CAAGPTDDTA YIDFEYF |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
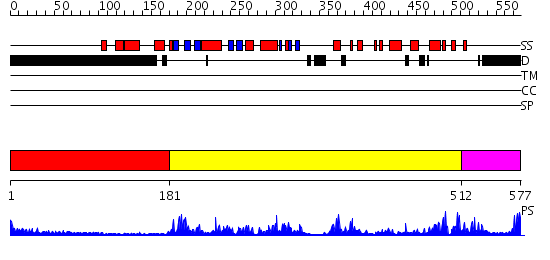
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..180] | N/A | No confident structure predictions are available. | |
| 2 | View Details | [181..511] | 64.522879 | Structure of human PDK1 kinase domain in complex with staurosporine | |
| 3 | View Details | [512..577] | 83.69897 | No description for 2jdoA was found. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)