













| General Information: |
|
| Name(s) found: |
klp-20 /
CE26143
[WormBase]
|
| Description(s) found:
SHOW ONLY BEST |
|
| Organism: | Caenorhabditis elegans |
| Length: | 646 amino acids |
Gene Ontology: |
|
| Cellular Component: |
microtubule associated complex
[IEA |
| Biological Process: |
microtubule-based movement
[IEA |
| Molecular Function: |
ATP binding
[IEA microtubule motor activity [IEA |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MEGAEKVKVV VRCRPISTTE KLQGHKIAVT CNDEEKAVNI KSLSQEDPPR TFYFDAVFSP 60
61 NTDQMTVYNV AARPIVENVL KGYNGTIFAY GQTGTGKTFT MAGDLEPVEM RGIIPNSFAH 120
121 IFDHIAKCQH DTTFLVRVSY LEIYNEEIRD LLSKDHNGNL EIKERPDVGV YVRNLSNPTV 180
181 ENASKMQALM EFGSKNRKVG ATAMNLESSR SHAMFTVTIE SCRNGLVTQG KLQLVDLAGS 240
241 ERQSKTGAQG ERLKEAAKIN LSLSTLGNVI SSLVDGKSTH IPYRNSKLTR LLQDSLGGNS 300
301 KTVMIANVGP ATYNYDETLS TLRYANRAKN IQNVAKINED PKDAQLRKFQ LEIEALRKIL 360
361 DEENPGDDEN QEEAWEAKMQ EREVEMEKKR KILEERVNSA VNDEETHRLV KEMMENEAEL 420
421 KKARSEHEKL RSKLEKIEKK LIVGGENLLE KVEEQAKLLE VNNKELEQSK FQEAHLRTQL 480
481 EERTAVKVEI EERYSSLQEE AFVKSKKIKK VSNELKDARA ELKDLEEDHQ RQVEAMLDDI 540
541 RQLRKELLLN IAIIDEYIPV EHVELIEKYV SWSEEHGDWQ LKAIAYTGNN MRASAPPAKK 600
601 EFSNNNQTVP MYYSYRADLG ASTAEHRPRT SSKKHRASIR LQQLLT |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
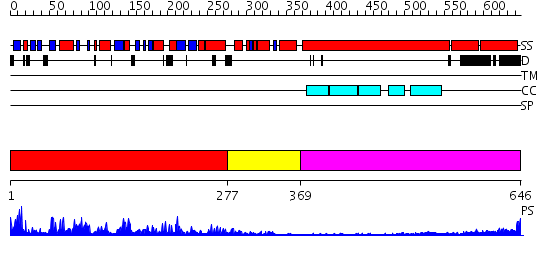
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..276] | 90.0 | Kinesin | |
| 2 | View Details | [277..368] | 10.69897 | KINESIN (DIMERIC) FROM RATTUS NORVEGICUS | |
| 3 | View Details | [369..646] | 4.30103 | Interferon-induced guanylate-binding protein 1 (GBP1), C-terminal domain; Interferon-induced guanylate-binding protein 1 (GBP1), N-terminal domain |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)