













| General Information: |
|
| Name(s) found: |
sck1 /
SPAC1B9.02c
[Sanger Pombe]
|
| Description(s) found:
Found 16 descriptions. SHOW ALL |
|
| Organism: | Schizosaccharomyces pombe |
| Length: | 696 amino acids |
Gene Ontology: |
|
| Cellular Component: |
cytosol
[IDA |
| Biological Process: |
protein amino acid phosphorylation
[TAS regulation of trehalose metabolic process [IMP cAMP-mediated signaling [IMP regulation of conjugation with cellular fusion [IGI |
| Molecular Function: |
ATP binding
[IC protein serine/threonine kinase activity [TAS |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MTEIFGKLHR SSNSENTNQA SPSTIQSHST QPVLSNDHST KVNDYEGKEG ASSNGYDPVF 60
61 MSDRMKMRYN EITAQLHKEQ SLKEDKESGS NSSESNGITP MGTYSEKPKL LQSRTPPSSC 120
121 YIRHDTVVPK DKNGQHAFGR LYVRLHQGRD LNVKSVNAQP YAVITFEKTQ VMVPPPFKDI 180
181 DGGIPISIPS KNRPLAGSAS GSSSGLHSEL MLADVRCPHW DFETVFDVTK MKSQMVVSVY 240
241 DKYEDDKFLG SVKITPIFLH EYVQEAWYKL EPLDLTKSLE GEIKVETIYE HIEHVRYGPE 300
301 DFTALRLIGK GTFGQVYLVR KNDTNRIYAM KKISKKLIVR KKEVTHTLGE RNILVRTSLD 360
361 ESPFIVGLKF SFQTASDLYL ITDYMSGGEL FWHLQHEGRF PEQRAKFYIA ELVLALEHLH 420
421 KHDIIYRDLK PENILLDADG HIALCDFGLS KANLSANATT NTFCGTTEYL APEVLLEDKG 480
481 YTKQVDFWSL GVLVFEMCCG WSPFYAPDVQ QMYRNIAFGK VRFPKGVLSS EGRSFVRGLL 540
541 NRNPNHRLGA VADTTELKEH PFFADINWDL LSKKKVQPPF KPNVQNDLDV SNFDKEFTNT 600
601 NVKNINIVSN VDPANASTPL SNTIQDRFRG FTFVNKSIDE QFQNLGLQEN EETDNLHACR 660
661 TTTHSSVNSI NSHGNPRTVD ANDPVADTVF GETFEA |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
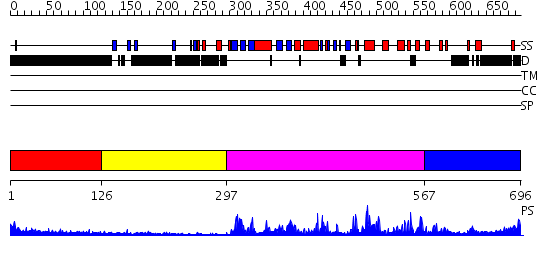
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..125] | N/A | No confident structure predictions are available. | |
| 2 | View Details | [126..296] | 5.91 | No description for 2ep6A was found. | |
| 3 | View Details | [297..566] | 70.045757 | 3-phosphoinositide dependent protein kinase-1 Pdk1 | |
| 4 | View Details | [567..696] | 95.69897 | Pkb kinase |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)