













| General Information: |
|
| Name(s) found: |
Mcm3-PA /
FBpp0070729
[FlyBase]
|
| Description(s) found:
Found 22 descriptions. SHOW ALL |
|
| Organism: | Drosophila melanogaster |
| Length: | 819 amino acids |
Gene Ontology: |
|
| Cellular Component: |
pre-replicative complex
[ISS][NAS]
|
| Biological Process: |
DNA replication initiation
[IEA]
DNA replication [IEA] pre-replicative complex assembly [ISS][NAS] |
| Molecular Function: |
DNA binding
[IEA]
ATP binding [IEA] chromatin binding [ISS][NAS] 3'-5' DNA helicase activity [IDA] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MAHEGEQFIK DIQREYVDFL DDEEDQGIYA GHVKDMIAEK SKRLIVNVND LKRKNPQRAL 60
61 GLLSNAADEQ LAFGRALKEY ASTVDPGYAK MHEDLFVGFE GCFGNRHVTP RSLTSIYLGN 120
121 MVCVEGIVTK VSLIRPKVVR SVHYCPNTRK VMERKYTDLT SFEAVPSGAA YPTKDEDGNL 180
181 LETEYGLSVY KDHQTLTIQE MPEKAPAGQL PRSVDIVCDD DLVDRCKPGD RVQIVGSYRC 240
241 LPGKRGGYTS GTFRTVLLAN NISLLSKESN LDISREDIML CKKLAKNNDI FELLSKSLAP 300
301 SIHGHAYVKQ AILCLLLGGV EKILPNGTRL RGDINVLLIG DPSVAKSQLL RYVLNTAPRA 360
361 IPTTGRGSSG VGLTAAVTTD QETGERRLEA GAMVLADRGV VCIDEFDKMS DIDRTAIHEV 420
421 MEQGRVTISK AGIHASLNAR CSVLAAANPV YGRYDQYKTP MENIGLQDSL LSRFDLLFVM 480
481 LDVIDSDVDQ MISDHVVRMH RYRNPKEADG EPLSMGSSYA DSLSFVSSSE EKKDTEVYEK 540
541 YDALLHGKSR QRHEKILSVE FMRKYIHIAK CMKPKLGEQA CEAIANEYSR LRSQEAVETD 600
601 VARTQPITAR TLETLIRLST AHARARMSKS VTIDDAHAAI ELVQFAYFKK VLDKDRPSKR 660
661 RRNSGSDAED DNGEASSQRS PSRRSKRTRT ATVGADSDEE DIEPPQPDAG DLTRRETRRS 720
721 LPARSVAMLM ASPSSEEQSV ATSTTEPAII SDARLGEFKN NLQRLFREAR EQSLALARIT 780
781 TAINVGSQEP FTAGEIEAAV HRMTEDNQIM VADDIVFLI |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
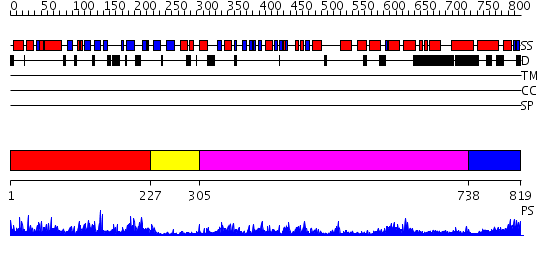
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..226] | 44.0 | DNA replication initiator (cdc21/cdc54) N-terminal domain | |
| 2 | View Details | [227..304] | 48.30103 | Crystal structure of sigm54 activator (AAA+ ATPase) in the inactive state | |
| 3 | View Details | [305..737] | 43.221849 | EDTA treated | |
| 4 | View Details | [738..819] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.98 |
Source: Reynolds et al. (2008)