













| General Information: |
|
| Name(s) found: |
PTK2_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 818 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MAGNGKDKEV DKSPSVSTLK LLGKRLFNSS SHTDNSSLLL SAEQLGNGRS LRKRPTSPSI 60
61 SGSGSGGNSP SSSAGARQRS ASLHRRKNNA SVGFSNGSVS SHKSSVALQD LIKHNNNPYL 120
121 NSPSDILGTG TGIASTRDRD RAVLDREKEK ERARNKERNT HHAGLPQRSN SMASHHFPNE 180
181 NIVYNPYGIS PNHARPDTAF ADTLNTNKEN DLSFYMHDGN SKIRMLPLPI ANPNDFLPED 240
241 MKQYSVHLTD NFVFDTDNKP IGSGGSSEVR KVKSSYRQKD VYALKKLNMI YHESPEKFYK 300
301 RCSKEFIIAK HLSHNVHITN TFYLLKVPTT TYTTRGWGFI MELGVKDLFQ LMERTGWKNV 360
361 PFNEKYCLFK QVAQGIKFCH DNGIAHRDLK PENVLISKEG ICKLTDFGIS DWYHVIPHDY 420
421 TSPVKTCQGM IGSPPYTPPE VMYFDAKKHY PEKFQKPYNP LAMDSYALGI MLITMINNII 480
481 PFIDSCNTDA RFREFEVSYD NFINHQNPHF RDKGCHKPGP GSEYSLARNF KNTDATRIAW 540
541 RLADPNPATR YTMDDLFNDP FFQQIETCVE PNDDDLVRVP ELRKSTSTND FSENSLDAPH 600
601 DQEVIHTSNP FLKKETLTSK PRSMLEIAES PSLKQKSKVK DSAKTKTHDV GDEGGNESTK 660
661 PKQQDKKENL KKDEVKNGDK DKVIEEATTT NVDSILEKPT PTSTKVEDNL SEDDSTMKEL 720
721 KSMLNSTPTT PTHNGPTPLP AKAGTQLDKR MSDLSLKSET PASTKNFSAP NVSSSSNSLR 780
781 SLGSPSVSSS KKKKVIHHHL DITNSVTNMS SVSAFISR |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
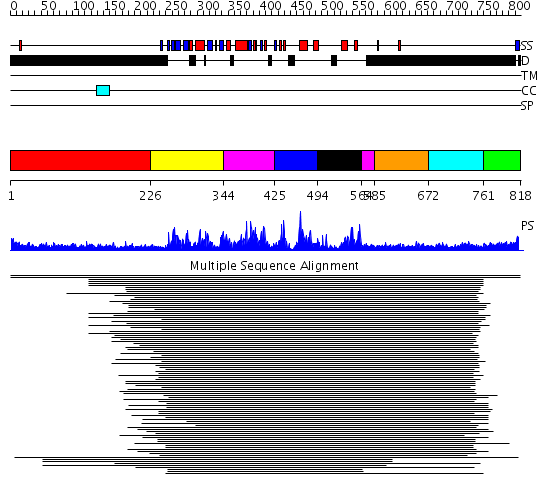
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..225] | N/A | No confident structure predictions are available. | |
| 2 | View Details | [226..343] | 256.9897 | Cyclin-dependent PK, CDK2 | |
| 3 | View Details | [344..424] [564..584] |
256.9897 | Cyclin-dependent PK, CDK2 | |
| 4 | View Details | [425..493] | 256.9897 | Cyclin-dependent PK, CDK2 | |
| 5 | View Details | [494..563] | 256.9897 | Cyclin-dependent PK, CDK2 | |
| 6 | View Details | [585..671] | 22.0 | Calmodulin | |
| 7 | View Details | [672..760] | 22.0 | Calmodulin | |
| 8 | View Details | [761..818] | N/A | Confident ab initio structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 8 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.97 |
Source: Reynolds et al. (2008)