













| General Information: |
|
| Name(s) found: |
FUN30_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 1131 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSGSHSNDED DVVQVPETSS PTKVASSSPL KPTSPTVPDA SVASLRSRFT FKPSDPSEGA 60
61 HTSKPLPSGS PEVALVNLAR EFPDFSQTLV QAVFKSNSFN LQSARERLTR LRQQRQNWTW 120
121 NKNASPKKSE TPPPVKKSLP LANTGRLSSI HGNINNKSSK ITVAKQKTSI FDRYSNVINQ 180
181 KQYTFELPTN LNIDSEALSK LPVNYNKKRR LVRADQHPIG KSYESSATQL GSAREKLLAN 240
241 RKYGRHANDN DEEEEESMMT DDDDASGDDY TESTPQINLD EQVLQFINDS DIVDLSDLSD 300
301 TTMHKAQLIA SHRPYSSLNA FVNTNFNDKD TEENASNKRK RRAAASANES ERLLDKITQS 360
361 IRGYNAIESV IKKCSSYGDL VTSQMKKWGV QVEGDNSELD LMNLGEDDDD DNDDGNNDNN 420
421 NSNNNNTAGA DATSKEKEDT KAVVEGFDET SAEPTPAPAP APVERETKRI RNTTKPKVVE 480
481 DEDDDVDLEA IDDELPQSEH EDDDYEEEDE DYNDEEEDVE YDDGDDDDDD DDEFVATRKN 540
541 THVISTTSRN GRKPIVKFFK GKPRLLSPEI SLKDYQQTGI NWLNLLYQNK MSCILADDMG 600
601 LGKTCQVISF FAYLKQINEP GPHLVVVPSS TLENWLREFQ KFAPALKIEP YYGSLQEREE 660
661 LRDILERNAG KYDVIVTTYN LAAGNKYDVS FLKNRNFNVV VYDEGHMLKN STSERFAKLM 720
721 KIRANFRLLL TGTPLQNNLK ELMSLLEFIM PNLFISKKES FDAIFKQRAK TTDDNKNHNP 780
781 LLAQEAITRA KTMMKPFILR RRKDQVLKHL PPKHTHIQYC ELNAIQKKIY DKEIQIVLEH 840
841 KRMIKDGELP KDAKEKSKLQ SSSSKNLIMA LRKASLHPLL FRNIYNDKII TKMSDAILDE 900
901 PAYAENGNKE YIKEDMSYMT DFELHKLCCN FPNTLSKYQL HNDEWMQSGK IDALKKLLKT 960
961 IIVDKQEKVL IFSLFTQVLD ILEMVLSTLD YKFLRLDGST QVNDRQLLID KFYEDKDIPI1020
1021 FILSTKAGGF GINLVCANNV IIFDQSFNPH DDRQAADRAH RVGQTKEVNI TTLITKDSIE1080
1081 EKIHQLAKNK LALDSYISED KKSQDVLESK VSDMLEDIIY DENSKPKGTK E |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
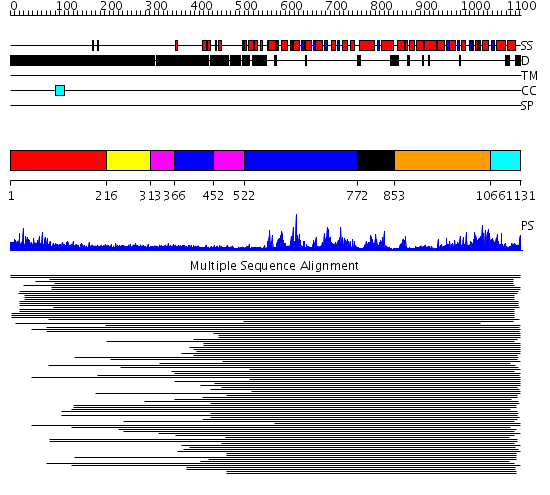
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..215] | 82.329997 | View MSA. No confident structure predictions are available. | |
| 2 | View Details | [216..312] | 5.522879 | RecG, N-terminal domain; RecG "wedge" domain; RecG helicase domain | |
| 3 | View Details | [313..365] [452..521] |
5.522879 | RecG, N-terminal domain; RecG "wedge" domain; RecG helicase domain | |
| 4 | View Details | [366..451] [522..771] |
5.522879 | RecG, N-terminal domain; RecG "wedge" domain; RecG helicase domain | |
| 5 | View Details | [772..852] | N/A | No confident structure predictions are available. | |
| 6 | View Details | [853..1065] | 7.0 | Putative DEAD box RNA helicase | |
| 7 | View Details | [1066..1131] | 5.0 | Nucleotide excision repair enzyme UvrB |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)