













| General Information: |
|
| Name(s) found: |
CDPKJ_ARATH
[Swiss-Prot]
|
| Description(s) found:
SHOW ONLY BEST |
|
| Organism: | Arabidopsis thaliana |
| Length: | 551 amino acids |
Gene Ontology: |
|
| Cellular Component: |
plasma membrane
[IDA]
|
| Biological Process: |
protein amino acid phosphorylation
[IEA][ISS]
|
| Molecular Function: |
ATP binding
[IEA]
calcium ion binding [IEA] protein kinase activity [IEA] calmodulin-dependent protein kinase activity [ISS] protein serine/threonine kinase activity [IEA] kinase activity [ISS] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MGCLCINLKK KVKKPTPDIS GEQNTEVKSR EITPKEQPRQ RQPAPRAKFQ IVVQPHKLPL 60
61 PLPQPQEKQK LINHQKQSTL QQPEPILGRP FEDIKEKYSL GRELGRGQFG ITYICTEISS 120
121 GKNFACKSIL KRKLIRTKDR EDVRREIQIM HYLSGQPNIV EIKGAYEDRQ SVHLVMELCE 180
181 GGELFDKITK RGHYSEKAAA EIIRSVVKVV QICHFMGVIH RDLKPENFLL SSKDEASSML 240
241 KATDFGVSVF IEEGKVYEDI VGSAYYVAPE VLKRNYGKAI DIWSAGVILY ILLCGNPPFW 300
301 AETDKGIFEE ILRGEIDFES EPWPSISESA KDLVRNMLKY DPKKRFTAAQ VLEHPWIREG 360
361 GEASDKPIDS AVLSRMKQLR AMNKLKKLAF KFIAQNLKEE ELKGLKTMFA NMDTDKSGTI 420
421 TYDELKSGLE KLGSRLTETE VKQLLEDADV DGNGTIDYIE FISATMNRFR VEREDNLFKA 480
481 FQHFDKDNSG FISRQELETA MKEYNMGDDI MIKEIISEVD ADNDGSINYQ EFCNMMKSCS 540
541 QSHQSKLVQP N |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
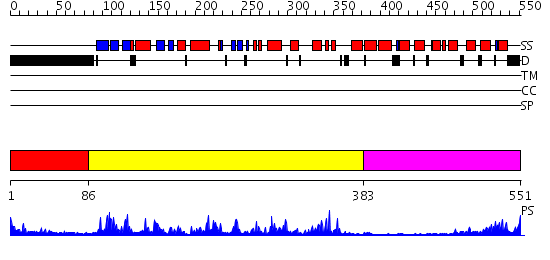
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..85] | N/A | No confident structure predictions are available. | |
| 2 | View Details | [86..382] | 77.39794 | CALMODULIN-DEPENDENT PROTEIN KINASE FROM RAT | |
| 3 | View Details | [383..551] | 23.69897 | Regulatory apparatus of Calcium Dependent protein kinase from Arabidopsis thaliana |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.94 |
Source: Reynolds et al. (2008)