













| General Information: |
|
| Name(s) found: |
Kif3C-PA /
FBpp0088260
[FlyBase]
|
| Description(s) found:
Found 19 descriptions. SHOW ALL |
|
| Organism: | Drosophila melanogaster |
| Length: | 649 amino acids |
Gene Ontology: |
|
| Cellular Component: |
kinesin complex
[ISS]
|
| Biological Process: |
microtubule-based movement
[IEA][ISS]
|
| Molecular Function: |
ATP binding
[IEA]
microtubule motor activity [ISS] minus-end-directed microtubule motor activity [NAS] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSENIKVVVR CRPMNQTEKE RNCQNIVEIN EFAVSVTNPS ARISQQKKFI FDSVYNMKTD 60
61 TEVIYDEMCY SLVESTIEGY NGTIFAYGQT GCGKTHTMQG DENFSNSTGI IPKCFDHIFE 120
121 RISMTTNVRY LALVTYLEIY NERIRDLLNK NENTNVINHF LKELPGIGVS VPTLTTQPVV 180
181 NANQCYDWLH FGNKNRVTAA TLMNKNSSRS HTIFTITLEQ SPFLNSIGSD AFGGICRGKL 240
241 SLVDLAGSER QRKTGAQGDR LKEASQINLS LSALGNVISS LVDGKAKHVP FRDSKLTRLL 300
301 QDSLGGNTKT LMISCISPTD IHYDETISTL RYASRAKNIS NKPKINEDPK DARLRQYQNE 360
361 ILYLKRMLQE SQQIINKNND PNKIIKSPLK IIQHTNMNST KNVQIIDLGR NCKASFKTNN 420
421 SILTKPNFPL IQSKEEVQLQ ARSRIDLIKR SLIGGERIHD FELKEKHMAR KYAAQRHLSA 480
481 IAIALSRVKC EDRDLLQGHY ATITQEIDIK NDYIRKCKEK IKMLEMEVSD LNSEFQLDRE 540
541 DYLDEIRNLG RQVKFHQQLF LKFSSNPGKF DRNWTPDTIL QNSLWNDDLK MWRIPESDLI 600
601 KLPPATQKSG LHCIHPLVIH SENQHNNQNE IVVGEKVSIS NIAFLFLSY |
SHOWING SINGLE HITS. [ Hide Single Hits ]
New Feature: Upload Your Own Microscopy Data
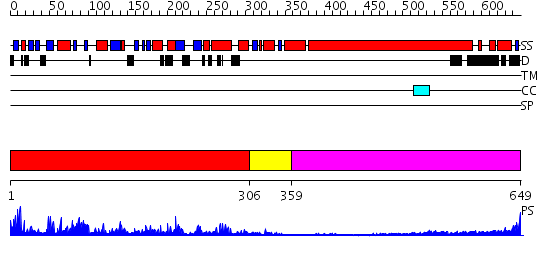
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..305] | 87.522879 | Kinesin | |
| 2 | View Details | [306..358] | 10.154902 | KINESIN (DIMERIC) FROM RATTUS NORVEGICUS | |
| 3 | View Details | [359..649] | 6.69897 | No description for 2i1jA was found. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)