













| General Information: |
|
| Name(s) found: |
gi|9758431
[NCBI NR]
gi|23506023 [NCBI NR] gi|16323188 [NCBI NR] gi|15240710 [NCBI NR] |
| Description(s) found:
Found 14 descriptions. SHOW ALL |
|
| Organism: | Arabidopsis thaliana |
| Length: | 613 amino acids |
Gene Ontology: |
|
| Cellular Component: |
nucleus
[IDA]
heterotrimeric G-protein complex [ISS] CUL4 RING ubiquitin ligase complex [ISS] |
| Biological Process: |
response to cold
[IMP]
histone deacetylation [IMP] |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSSLTSVELN FLVFRYLQES GFTHAAFTLG YEAGINKSNI DGNMVPPGAL IKFVQKGLQY 60
61 MEMEANLSNS EVDIDEDFSF FQPLDLISKD VKELQDMLRE KKRKERDMEK ERDRSKENDK 120
121 GVEREHEGDR NRAKEKDRHE KQKERERERE KLEREKERER EKIEREKERE REKMEREIFE 180
181 REKDRLKLEK EREIERERER EKIEREKSHE KQLGDADREM VIDQTDKEIA GDGSTGAEPM 240
241 DIVMTPTSQT SHIPNSDVRI LEGHTSEVCA CAWSPSASLL ASGSGDATAR IWSIPEGSFK 300
301 AVHTGRNINA LILKHAKGKS NEKSKDVTTL DWNGEGTLLA TGSCDGQARI WTLNGELIST 360
361 LSKHKGPIFS LKWNKKGDYL LTGSVDRTAV VWDVKAEEWK QQFEFHSGPT LDVDWRNNVS 420
421 FATSSTDSMI YLCKIGETRP AKTFTGHQGE VNCVKWDPTG SLLASCSDDS TAKIWNIKQS 480
481 TFVHDLREHT KEIYTIRWSP TGPGTNNPNK QLTLASASFD STVKLWDAEL GKMLCSFNGH 540
541 REPVYSLAFS PNGEYIASGS LDKSIHIWSI KEGKIVKTYT GNGGIFEVCW NKEGNKIAAC 600
601 FADNSVCVLD FRM |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
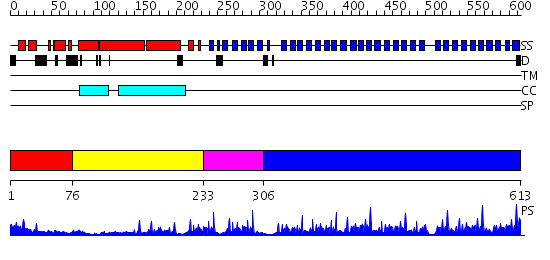
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..75] | 9.0 | N-terminal domain of Lissencephaly-1 protein (Lis-1) | |
| 2 | View Details | [76..232] | 1.6 | crystal structure of the cytotoxic bacterial protein colicin B at 2.5 A resolution | |
| 3 | View Details | [233..305] | 37.39794 | Crystal Structure Of The C-Terminal WD40 Domain Of Sif2 | |
| 4 | View Details | [306..613] | 84.522879 | PAF-AH Holoenzyme: Lis1/Alfa2 |
Functions predicted (by domain):
| # | Gene Ontology predictions | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.97 |
Source: Reynolds et al. (2008)