













| General Information: |
|
| Name(s) found: |
MCM5_ARATH
[Swiss-Prot]
|
| Description(s) found:
Found 12 descriptions. SHOW ALL |
|
| Organism: | Arabidopsis thaliana |
| Length: | 727 amino acids |
Gene Ontology: |
|
| Cellular Component: |
nucleus
[IDA]
cytoplasm [IDA] |
| Biological Process: |
DNA replication initiation
[ISS]
DNA unwinding involved in replication [TAS] |
| Molecular Function: |
DNA binding
[IEA][ISS]
ATP binding [IEA][ISS] DNA-dependent ATPase activity [ISS] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSGWDEGAVY YSDQPQFPEA GDAATISPHA VLTKFKEFIR NFEIEQNCFP YREALLDNPK 60
61 RLVVHLEDLL SFDSDLPSLI RSAPADYLPV FEKAAGEVLT GLKMREANEG GVMEEPLTRD 120
121 VQILLTSRED PVSMRLLGAQ YISKLVKISG ISIAASRVKA KATYVFLVCK NCKKTREVPC 180
181 RPGLGGAIVP RSCDNIPQPG EEPCPLDPWM VVPDRSQYVD QQTLKLQENP EDVPTGELPR 240
241 NMLLSVDRHL VQTIVPGTRL TVMGIYSIFQ ASSSSNSHKG AVAIRQPYIR VVGLEDTNEA 300
301 SSRGPANFTP DEEEEFKKFA DSQDVYKNIC TKIAPSIFGH EDVKRAAACL LFGGSRKSLP 360
361 DGVKLRGDIN VLLLGDPSTA KSQFLKFVEK TAPIAVYTSG KGSSAAGLTA SVIRDSSTRE 420
421 FYLEGGAMVL ADGGVVCIDE FDKMRPEDRV AIHEAMEQQT ISIAKAGITT VLNSRTSVLA 480
481 AANPPSGRYD DLKTAQDNID LQTTILSRFD LIFIVKDIRK YSQDKEIASH IIRVHASANK 540
541 FSDENTDSKE DNWLKRYIQY CRARCHPRLS KDAAENLQRK YVTIRMDMKR RAHETGEAAP 600
601 IPITVRQLEA IVRLSESLAK MRLSHEATPD DVDKAFKLFD TSTMDAARSG INQQINITGE 660
661 MANEIKQAET QIKRRMGIGA RLSERRLIED LARMGMNDSM VRRALLIMHQ RGEVEYQRER 720
721 RSIVRKA |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
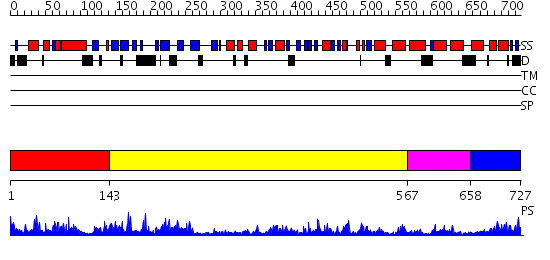
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..142] | 44.522879 | DNA replication initiator (cdc21/cdc54) N-terminal domain | |
| 2 | View Details | [143..566] | 16.221849 | N-terminal, ClpS-binding domain of ClpA, an Hsp100 chaperone; ClpA, an Hsp100 chaperone, AAA+ modules | |
| 3 | View Details | [567..657] | 14.0 | Holliday junction helicase RuvB | |
| 4 | View Details | [658..727] | 50.0 | Crystal structure of a sigma54-activator suggests the mechanism for the conformational switch necessary for sigma54 binding |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.97 |
Source: Reynolds et al. (2008)