













| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MATRGPTPDK ARMGLPDRWL HCPKTGTLIN NLFFPFKTPL CKMYDNQIAE RRYQFHPAEV 60
61 FSHPHLHGKK IGLWIDLTNT DRYYFREEVT EHECIYHKMK MAGRGVSPTQ EDTDNFIKLV 120
121 QEFHKKYPDR VVGVHCTHGF NRTGFLIAAY LFQVEEYGLD AAIGEFAENR QKGIYKQDYI 180
181 DDLFARYDPT EDDKILAPEK PDWEREMSIG MSTQIDNGRP STSQQIPATN GNNNQNGNQL 240
241 SGGGDNSKLF MDGLIRGVKV CEDEGKKSML QAKIKNLCKY NKQGFPGLQP VSLSRGNINL 300
301 LEQESYMVSW KADGMRYIIY INDGDVYAFD RDNEVFEIEN LDFVTKNGAP LMETLVDTEV 360
361 IIDKVEINGA MCDQPRMLIY DIMRFNSVNV MKEPFYKRFE IIKTEIIDMR TAAFKTGRLK 420
421 HENQIMSVRR KDFYDLEATA KLFGPKFVQH VGHEIDGLIF QPKKTKYETG RCDKVLKWKP 480
481 PSHNSVDFLL KVEKKCKEGM LPEWIGYLFV QNLSDPFGTM KATATLKKYH NKIIECTLLV 540
541 DNQGRPKEWK FMRERTDKSL PNGLRTAENV VETMVNPVTE TYLIEYVNHA LRVLKRAAAA 600
601 HRHHQIHQQQ LHEGEPEARR QKL |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
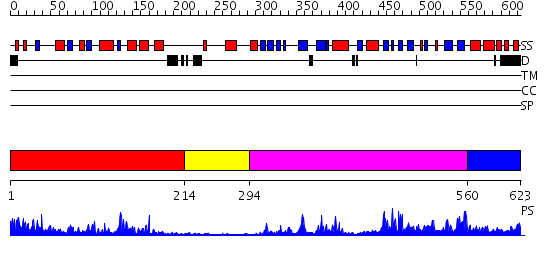
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..213] | 34.30103 | CRYSTAL STRUCTURE OF THE HUMAN RNA guanylyltransferase and 5'- phosphatase | |
| 2 | View Details | [214..293] | 1.0 | mRNA capping enzyme alpha subunit | |
| 3 | View Details | [294..559] | 13.0 | New Crystal Structure of Chlorella Virus DNA Ligase-Adenylate | |
| 4 | View Details | [560..623] | 42.221849 | mRNA capping enzyme alpha subunit |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 |
|
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)