













| General Information: |
|
| Name(s) found: |
gi|71153821
[NCBI NR]
gi|24640536 [NCBI NR] gi|22831933 [NCBI NR] gi|15554294 [NCBI NR] |
| Description(s) found:
Found 12 descriptions. SHOW ALL |
|
| Organism: | Drosophila melanogaster |
| Length: | 1161 amino acids |
Gene Ontology: |
|
| Cellular Component: |
cellular_component
[ND]
|
| Biological Process: |
dorsal closure
[IMP][TAS]
imaginal disc fusion, thorax closure [IMP] positive regulation of JNK cascade [IMP] dorsal closure, elongation of leading edge cells [IMP] activation of JNKK activity [TAS] protein amino acid autophosphorylation [ISS] activation of JUN kinase activity [IMP][TAS] dorsal appendage formation [IMP] JNK cascade [IGI][IMP] protein amino acid phosphorylation [IDA][NAS] imaginal disc-derived female genitalia morphogenesis [IMP] imaginal disc-derived male genitalia morphogenesis [IMP] |
| Molecular Function: |
ATP binding
[IEA]
protein tyrosine kinase activity [IEA] protein serine/threonine/tyrosine kinase activity [ISS] protein homodimerization activity [ISS] JUN kinase kinase kinase activity [IDA][ISS][NAS] protein kinase activity [IDA][NAS] |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MPSQVSRGLD TTNMLPISEE QQLQQQQQQQ QLEQLHHPQI PEIPIPDLEQ VETQVGDGSL 60
61 WTALYDYDAQ GEDELTLRRG EIVVVLSTDS EVSGDVGWWT GKIGDKVGVF PKDFVTDEDP 120
121 LQLNVSSAIG DIQPHEIEYN ELDIKEVIGS GGFCKVHRGY YDGEEVAIKI AHQTGEDDMQ 180
181 RMRDNVLQEA KLFWALKHEN IAALRGVCLN TKLCLVMEYA RGGSLNRILA GKIPPDVLVN 240
241 WAIQIARGMN YLHNEAPMSI IHRDLKSSNV LIYEAIEGNH LQQKTLKITD FGLAREMYNT 300
301 QRMSAAGTYA WMPPEVISVS TYSKFSDVWS YGVLLWELIT GETPYKGFDP LSVAYGVAVN 360
361 TLTLPIPKTC PETWGALMKS CWQTDPHKRP GFKEILKQLE SIACSKFTLT PQESFHYMQE 420
421 CWRKEIAGVL HDLREKEKEL RNKEEQLLRV QNEQREKANL LKIREQNLRE RERVLIEREL 480
481 VMLQPVPSKR KHKKGKKNKP LQISLPTGFR HTITAVRDKA EQPGSPSFSG LRIVALTDGH 540
541 KGKTWGPSTM HQRERSLLPS QLSGGQPEWP AQTSTHSSFS KSAPNLDKKQ QQQNQQQVAS 600
601 LTPPPGLGIL GGSGGAGGTP ATPLLYPGIP IILTRPNNNN IGNCKAITTT ITTTTTTTTN 660
661 NNNNNNNSIS ANNNNQLNNI STINSNNNNN QTNLTSQPNT IIVLQNGRNN SNSSTTSQSP 720
721 AKIYHRARSQ EYGLDHPLAY QPPPLYLVTD DSSETDTVAS PTGCFHFLKS GNSSAASGAV 780
781 HLHRFGGSLG NSPAVGRKKH SLDSSSHHPP ANGSNSFALP NQLTLPSEDN NTYDHAFYRD 840
841 VIKKMSMASS ERVNSKSSGD LTMYNSSTPL TARDCDDAEE AFEGGRFQRN FSGSQFPRHC 900
901 FFTRQEEEGE AEDEDAVAAE VDTADADADD ECQVPASQMR QNSTTSRKSS VTFQSVSFEE 960
961 PDFVATPRTT ARSDLYTSSA SISFATYRSA SPSLSSSSTT ASASPSIAST EAVNGYHMQE1020
1021 NSILNTRRMQ DVQPHPDVIK LRAQEQRQQT KNQKKQRPKH ITKSKSVEAP VEGQHHEHDD1080
1081 HNDPQHQHHS AGSSKIRALF NLFTRSRKKY SKLAEHNMVG GPEFCAIDPY QTDLAMGGSS1140
1141 RSLKRKGKKP QTQSCEQLER C |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
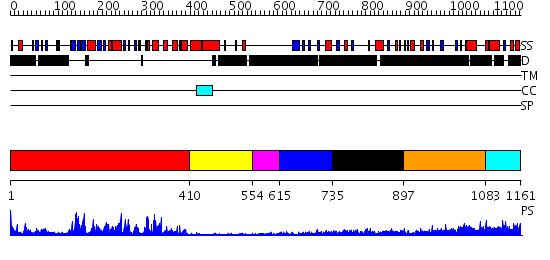
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..409] | 92.39794 | CHICKEN SRC TYROSINE KINASE | |
| 2 | View Details | [410..553] | 48.0 | TWITCHIN KINASE FRAGMENT (C.ELEGANS), AUTOREGULATED PROTEIN KINASE AND IMMUNOGLOBULIN DOMAINS | |
| 3 | View Details | [554..614] | N/A | No confident structure predictions are available. | |
| 4 | View Details | [615..734] | N/A | No confident structure predictions are available. | |
| 5 | View Details | [735..896] | N/A | No confident structure predictions are available. | |
| 6 | View Details | [897..1082] | 2.279994 | View MSA. No confident structure predictions are available. | |
| 7 | View Details | [1083..1161] | N/A | No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)