













| General Information: |
|
| Name(s) found: |
MCM5
[HGNC (HUGO)]
|
| Description(s) found:
Found 41 descriptions. SHOW ALL |
|
| Organism: | Homo sapiens |
| Length: | 734 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSGFDDPGIF YSDSFGGDAQ ADEGQARKSQ LQRRFKEFLR QYRVGTDRTG FTFKYRDELK 60
61 RHYNLGEYWI EVEMEDLASF DEDLADYLYK QPAEHLQLLE EAAKEVADEV TRPRPSGEEV 120
121 LQDIQVMLKS DASPSSIRSL KSDMMSHLVK IPGIIIAASA VRAKATRISI QCRSCRNTLT 180
181 NIAMRPGLEG YALPRKCNTD QAGRPKCPLD PYFIMPDKCK CVDFQTLKLQ ELPDAVPHGE 240
241 MPRHMQLYCD RYLCDKVVPG NRVTIMGIYS IKKFGLTTSR GRDRVGVGIR SSYIRVLGIQ 300
301 VDTDGSGRSF AGAVSPQEEE EFRRLAALPN VYEVISKSIA PSIFGGTDMK KAIACLLFGG 360
361 SRKRLPDGLT RRGDINLLML GDPGTAKSQL LKFVEKCSPI GVYTSGKGSS AAGLTASVMR 420
421 DPSSRNFIME GGAMVLADGG VVCIDEFDKM REDDRVAIHE AMEQQTISIA KAGITTTLNS 480
481 RCSVLAAANS VFGRWDETKG EDNIDFMPTI LSRFDMIFIV KDEHNEERDV MLAKHVITLH 540
541 VSALTQTQAV EGEIDLAKLK KFIAYCRVKC GPRLSAEAAE KLKNRYIIMR SGARQHERDS 600
601 DRRSSIPITV RQLEAIVRIA EALSKMKLQP FATEADVEEA LRLFQVSTLD AALSGTLSGV 660
661 EGFTSQEDQE MLSRIEKQLK RRFAIGSQVS EHSIIKDFTK QKYPEHAIHK VLQLMLRRGE 720
721 IQHRMQRKVL YRLK |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
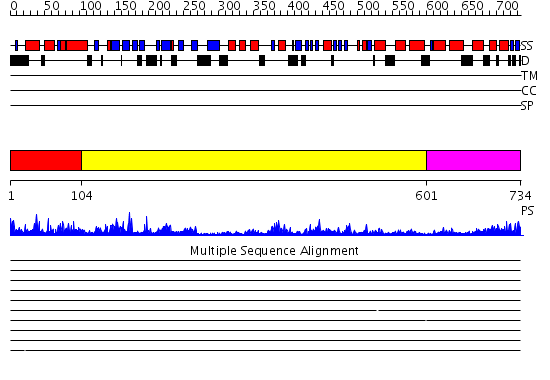
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..103] | 35.39794 | DNA replication initiator (cdc21/cdc54) N-terminal domain | |
| 2 | View Details | [104..600] | 40.69897 | N-terminal, ClpS-binding domain of ClpA, an Hsp100 chaperone; ClpA, an Hsp100 chaperone, AAA+ modules | |
| 3 | View Details | [601..734] | 35.69897 | EDTA treated |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.93 |
Source: Reynolds et al. (2008)