













| General Information: |
|
| Name(s) found: |
MCM2_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 868 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MSDNRRRRRE EDDSDSENEL PPSSPQQHFR GGMNPVSSPI GSPDMINPEG DDNEVDDVPD 60
61 IDEVEEQMNE VDLMDDNMYE DYAADHNRDR YDPDQVDDRE QQELSLSERR RIDAQLNERD 120
121 RLLRNVAYID DEDEEQEGAA QLDEMGLPVQ RRRRRRQYED LENSDDDLLS DMDIDPLREE 180
181 LTLESLSNVK ANSYSEWITQ PNVSRTIARE LKSFLLEYTD ETGRSVYGAR IRTLGEMNSE 240
241 SLEVNYRHLA ESKAILALFL AKCPEEMLKI FDLVAMEATE LHYPDYARIH SEIHVRISDF 300
301 PTIYSLRELR ESNLSSLVRV TGVVTRRTGV FPQLKYVKFN CLKCGSILGP FFQDSNEEIR 360
361 ISFCTNCKSK GPFRVNGEKT VYRNYQRVTL QEAPGTVPPG RLPRHREVIL LADLVDVSKP 420
421 GEEVEVTGIY KNNYDGNLNA KNGFPVFATI IEANSIKRRE GNTANEGEEG LDVFSWTEEE 480
481 EREFRKISRD RGIIDKIISS MAPSIYGHRD IKTAVACSLF GGVPKNVNGK HSIRGDINVL 540
541 LLGDPGTAKS QILKYVEKTA HRAVFATGQG ASAVGLTASV RKDPITKEWT LEGGALVLAD 600
601 KGVCLIDEFD KMNDQDRTSI HEAMEQQSIS ISKAGIVTTL QARCSIIAAA NPNGGRYNST 660
661 LPLAQNVSLT EPILSRFDIL CVVRDLVDEE ADERLATFVV DSHVRSHPEN DEDREGEELK 720
721 NNGESAIEQG EDEINEQLNA RQRRLQRQRK KEEEISPIPQ ELLMKYIHYA RTKIYPKLHQ 780
781 MDMDKVSRVY ADLRRESIST GSFPITVRHL ESILRIAESF AKMRLSEFVS SYDLDRAIKV 840
841 VVDSFVDAQK VSVRRQLRRS FAIYTLGH |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
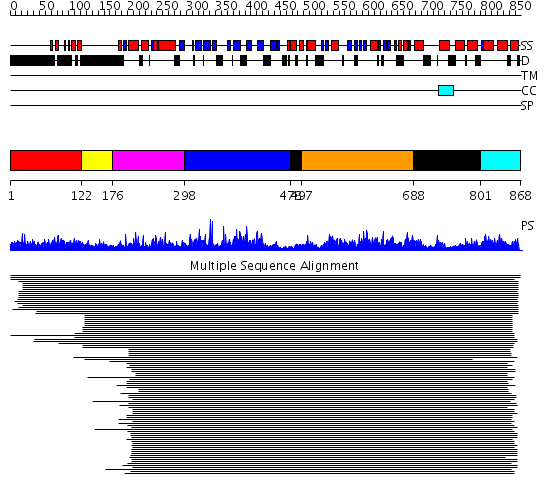
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..121] | 1.015984 | View MSA. No confident structure predictions are available. | |
| 2 | View Details | [122..175] | N/A | No confident structure predictions are available. | |
| 3 | View Details | [176..297] | 1.095989 | View MSA. Confident ab initio structure predictions are available. | |
| 4 | View Details | [298..477] | 7.221849 | MCM2/3/5 family No confident structure predictions are available. | |
| 5 | View Details | [478..496] [688..800] |
2.522879 | Replication factor C | |
| 6 | View Details | [497..687] | 2.522879 | Replication factor C | |
| 7 | View Details | [801..868] | 1.494987 | View MSA. No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 7 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)