













| General Information: |
|
| Name(s) found: |
ISW2_YEAST
[Swiss-Prot]
|
| Description(s) found: |
|
| Organism: | Saccharomyces cerevisiae S288c |
| Length: | 1120 amino acids |
Gene Ontology: |
|
| Cellular Component: | NONE FOUND |
| Biological Process: | NONE FOUND |
| Molecular Function: | NONE FOUND |
| Sequence: |
|
| Sequence: [PDR BLAST] [ProtParam] |
1 11 21 31 41 51
| | | | | |
1 MTTQQEEQRS DTKNSKSESP SEVLVDTLDS KSNGSSDDDN IGQSEELSDK EIYTVEDRPP 60
61 EYWAQRKKKF VLDVDPKYAK QKDKSDTYKR FKYLLGVTDL FRHFIGIKAK HDKNIQKLLK 120
121 QLDSDANKLS KSHSTVSSSS RHHRKTEKEE DAELMADEEE EIVDTYQEDI FVSESPSFVK 180
181 SGKLRDYQVQ GLNWLISLHE NKLSGILADE MGLGKTLQTI SFLGYLRYVK QIEGPFLIIV 240
241 PKSTLDNWRR EFLKWTPNVN VLVLHGDKDT RADIVRNIIL EARFDVLITS YEMVIREKNA 300
301 LKRLAWQYIV IDEAHRIKNE QSALSQIIRL FYSKNRLLIT GTPLQNNLHE LWALLNFLLP 360
361 DIFGDSELFD EWFEQNNSEQ DQEIVIQQLH SVLNPFLLRR VKADVEKSLL PKIETNVYVG 420
421 MTDMQIQWYK SLLEKDIDAV NGAVGKREGK TRLLNIVMQL RKCCNHPYLF EGAEPGPPYT 480
481 TDEHLIFNSG KMIILDKLLK RLKEKGSRVL IFSQMSRLLD ILEDYCYFRD FEYCRIDGST 540
541 SHEERIEAID EYNKPNSEKF VFLLTTRAGG LGINLVTADT VILFDSDWNP QADLQAMDRA 600
601 HRIGQKKQVH VYRFVTENAI EEKVIERAAQ KLRLDQLVIQ QGTGKKTASL GNSKDDLLDM 660
661 IQFGAKNMFE KKASKVTVDA DIDDILKKGE QKTQELNAKY QSLGLDDLQK FNGIENQSAY 720
721 EWNGKSFQKK SNDKVVEWIN PSRRERRREQ TTYSVDDYYK EIIGGGSKSA SKQTPQPKAP 780
781 RAPKVIHGQD FQFFPKELDA LQEKEQLYFK KKVNYKVTSY DITGDIRNEG SDAEEEEGEY 840
841 KNAANTEGHK GHEELKRRIE EEQEKINSAP DFTQEDELRK QELISKAFTN WNKRDFMAFI 900
901 NACAKYGRDD MENIKKSIDS KTPEEVEVYA KIFWERLKEI NGWEKYLHNV ELGEKKNEKL 960
961 KFQETLLRQK IEQCKHPLHE LIIQYPPNNA RRTYNTLEDK FLLLAVNKYG LRADKLYEKL1020
1021 KQEIMMSDLF TFDWFIKTRT VHELSKRVHT LLTLIVREYE QPDANKKKRS RTSATREDTP1080
1081 LSQNESTRAS TVPNLPTTMV TNQKDTNDHV DKRTKIDQEA |
NOT SHOWING SINGLE HITS. [ Show Single Hits ]
New Feature: Upload Your Own Microscopy Data
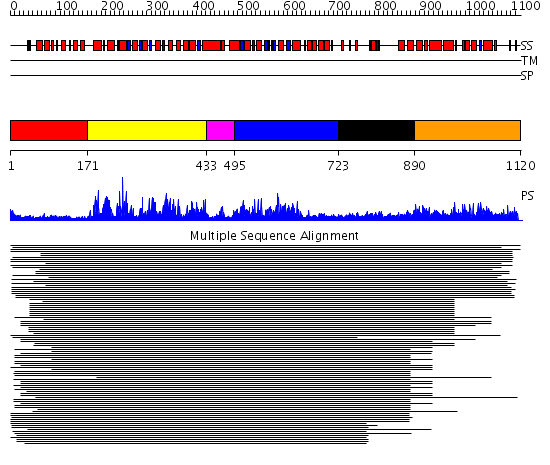
Domains predicted:
| # | Region(s) | Method | Confidence | Match Description | |
| 1 | View Details | [1..170] | 1.482995 | View MSA. No confident structure predictions are available. | |
| 2 | View Details | [171..432] | 105.29243 | SNF2 family N-terminal domain No confident structure predictions are available. | |
| 3 | View Details | [433..494] | 4.522879 | Putative DEAD box RNA helicase | |
| 4 | View Details | [495..722] | 2.69897 | Nucleotide excision repair enzyme UvrB | |
| 5 | View Details | [723..889] | 2.211994 | View MSA. No confident structure predictions are available. | |
| 6 | View Details | [890..1120] | 1.066996 | View MSA. No confident structure predictions are available. |
Functions predicted (by domain):
| # | Gene Ontology predictions | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 1 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 3 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 4 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 5 | No functions predicted. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 6 | No functions predicted. |
| Protein predicted to be: | GLOBULAR (No transmembrane regions or signal peptide) |
| Confidence of classification: | 0.99 |
Source: Reynolds et al. (2008)